Joint Subproject 4
Steigerung der Pandemievorbereitung durch computergestützte Hochdurchsatz-Virusentdeckung: Identifikation von RNA-Viren und Wirtsreservoirs mit hohem Spillover-Risiko
Wir haben ein langjähriges Interesse an der Entdeckung und Evolution von Viren im Allgemeinen. Unsere Strategie umfasst zwei computergestützte Pipelines, genannt Virushunter und Virusgatherer, die jeweils der Identifikation von NGS-Experimenten dienen, die positiv auf das Vorhandensein von viralen Sequenzreads sind, und der Assemblierung der entsprechenden viralen Genome.
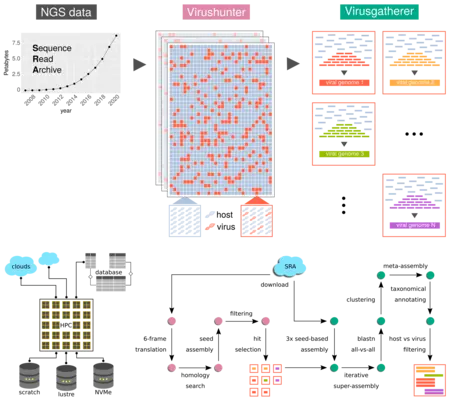
In dem vorgeschlagenen Projekt werden wir nun diese Daten nutzen, um sowohl zu nachhaltigen Strategien für die Vorbereitung auf zukünftige aufkommende Infektionskrankheiten (EIDs) und Pandemien beizutragen als auch um die aktuelle Pandemie, die durch SARS-CoV-2 verursacht wird, besser zu verstehen und zu kontrollieren. Im Hinblick auf die Vorbereitung auf zukünftige EIDs werden wir einen Spillover- und Pandemierisiko-Score ableiten, validieren und anwenden, um RNA-Viren mit der höchsten Zoonose-Wahrscheinlichkeit zu identifizieren. Zur Kontrolle der aktuellen Pandemie werden wir einen ACE2-Rezeptor-Sequenzkatalog erstellen, um potenzielle Tierreservoirs von SARS-CoV-2 im Besonderen und von viralen EIDs im Allgemeinen zu identifizieren. Weiterhin werden wir die mögliche Kreuzinterferenz zwischen SARS-CoV-2 und anderen Atemwegsviren sowie allgegenwärtigen persistenten Viren untersuchen.




