Knowledge Modelling

Dr. Wahyu Wijaya Hadiwikarta
Leiter Knowledge Modelling & RadPlanBio
In der Knowledge Modeling-Arbeitsgruppe entwickeln wir Methoden zur Wissensextraktion aus verknüpften präklinischen und klinischen Strahlentherapie-Studien. Diese Datensätze sind aufgrund struktureller Komplexität und multimodaler Eigenschaften heterogen. Zur Bewältigung dieser Herausforderungen entwerfen wir Modelle, die stochastische Variabilität in einem semantisch verknüpften Wissensgraphen erfassen und eine robuste, interpretierbare Datenintegration ermöglichen.

Unsere Forschung
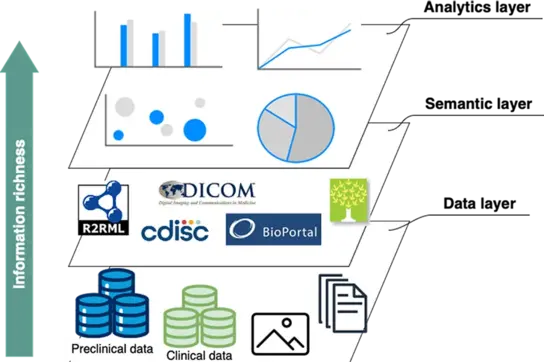
Wir wenden semantische Methoden an, um präklinische und klinische Datensätze in Wissensgraphen (sog. knowledge graphs, KG) zu integrieren und so eine strukturierte, interoperable Repräsentation biomedizinischer Informationen zu schaffen. Durch die Nutzung domänenspezifischer Ontologien verbessern wir die Datenstandardisierung und gewährleisten die Einhaltung der FAIR-Prinzipien (Findable, Accessible, Interoperable, Reusable), was die Wissensentdeckung erleichtert und die translationale Forschung unterstützt. Unser Ansatz wird auf Datensätze aus mehreren Studien angewendet, die auf der RadPlanBio-Plattform des Deutschen Krebskonsortiums (DKTK) gehostet werden, wo wir Daten aus präklinischen und klinischen Bereichen systematisch organisieren und verknüpfen. Präklinische Datensätze entsprechen dem SEND-Standard für die tabellarische Erfassung nichtklinischer Daten und werden mit der PTRO-Ontologie angereichert, die derzeit aktiv weiterentwickelt wird. Die Erweiterung dieser FAIRifizierung auf klinische Daten wird die Datenintegration, Zugänglichkeit und Nutzbarkeit weiter verbessern und somit Fortschritte in der personalisierten Medizin vorantreiben sowie Innovationen in der therapeutischen Entwicklung fördern.
Um tiefere Einblicke zu gewinnen, entwickeln wir analytische Methoden auf Basis des Propagator-Konzepts der Quantenmechanik. Dabei wollen wir die strukturierte Repräsentation von Wissen nutzen, um die Mechanismen der Krebsprogression zu erschließen. Der vollständige Satz numerischer und nicht-numerischer Daten, der einem Patienten zu einem bestimmten Zeitpunkt zugeordnet ist, liefert eine Momentaufnahme seines Zustands – eine abstrakte „Position“ im Raum aller möglichen Krankheitsverläufe. Im Zeitverlauf verändert sich dieser Zustand beobachtbar, was wir als durch eine Transferfunktion, den sogenannten Propagator, beschreibbar annehmen. Dieser Ansatz ermöglicht eine systematische Modellierung der Krankheitsdynamik, bietet eine methodisch fundierte Möglichkeit zur Vorhersage von Krankheitsverläufen und hilft, kritische Übergänge in der Krebsprogression zu identifizieren.
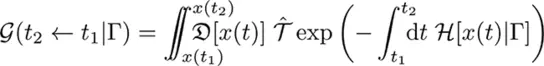
Basierend auf den beobachteten Daten über den Zustand einer:s Proband:in/en zu bestimmten Zeitpunkten und einem Regelsatz (Γ) für die Entwicklung, berücksichtigt der Propagator (G) alle möglichen Pfade, die die/der Proband:in unter Berücksichtigung unseres begrenzten Wissens hätte nehmen können.
Durch die gezielte Anpassung der zugrundeliegenden Regeln des Propagators und die Schätzung ihrer Wahrscheinlichkeit anhand unserer Daten versuchen wir, die wahrscheinlichsten Mechanismen des Krebswachstums zu ermitteln. Mit diesen Regeln möchten wir das Tumorwachstum und die Überlebenswahrscheinlichkeit besser verstehen und vorhersagen, um letztendlich fundierte Entscheidungen zur Behandlungsstrategie zu unterstützen.
Kontaktieren Sie uns

Dr. Wahyu Wijaya Hadiwikarta
Leiter Knowledge Modelling & RadPlanBio