Präzisions-Sarkomforschung
- Funktionelle und Strukturelle Genomforschung
- NCT
- Nachwuchsgruppe

Dr. Priya Chudasama
Group leader
Wir erforschen molekulare und klinische Merkmale von Sarkomen mithilfe struktureller und funktioneller Genomikansätze, um die Sarkomgenese besser zu verstehen und präzisere, wirksamere sowie gut verträgliche Therapien für Sarkom-Patienten zu entwickeln.

Unsere Forschung
Sarkome werden hauptsächlich in zwei Arten unterteilt: Knochensarkome und Weichteilsarkome. Sarkome weisen eine bemerkenswerte genetische und histologische Vielfalt auf, die sich in mehr als 150 Subtypen nach der Klassifikation der Weltgesundheitsorganisation widerspiegelt, was wiederum erhebliche diagnostische und therapeutische Schwierigkeiten mit sich bringt. „Handlungsfähige“ Läsionen, die eine Vorhersage des Ansprechens auf konventionelle oder zielgerichtete Krebsmedikamente ermöglichen und/oder direkte Angriffspunkte für therapeutische Maßnahmen darstellen, fehlen in den meisten Fällen, da die Ereignisse, die die Sarkomentwicklung vorantreiben, nur unvollständig verstanden werden.
Die vom Emmy Noether-Programm der Deutschen Forschungsgemeinschaft (DFG) geförderte Arbeitsgruppe „Precision Sarcoma Research“ wurde mit dem Ziel gegründet, die der Tumorentwicklung zugrunde liegenden molekularen Veränderungen besser zu verstehen und neue Zielstrukturen für eine präzise Krebstherapie zu identifizieren. Zu diesem Zweck nutzen wir Tumor-Multiomics-Daten, die im Rahmen der internen Präzisions-Onkologie-Workflows, insbesondere des NCT/Deutschen Krebskonsortiums (DKTK) MASTER (Molecularly Aided Stratification for Tumor Eradication), generiert wurden, sowie öffentlich zugängliche Datenressourcen, um die genomischen, epigenomischen, transkriptomischen und immunologischen Landschaften von Sarkomen systematisch zu untersuchen, um pan-sarkom- oder subentitätsspezifische pathognomonische Veränderungen zu identifizieren. Projektspezifisch haben wir am DKFZ neueste Technologieplattformen wie Long-Read-Sequenzierung, Einzelkernsequenzierung sowie Spatial Transcriptomics eingesetzt, um tiefere Einblicke in die Biologie der Tumoren zu gewinnen. Mechanistische Untersuchungen ausgewählter Aberrationen werden durch unser wachsendes Modellsystem-Panel und ein umfassendes Toolkit für funktionelle Genomik-Untersuchungen (z.B. CRISPR/Cas9-Bibliotheken) ermöglicht.
Gegenwärtig verfolgen wir die folgenden Ziele:
1. Ausrichtung auf kritische Krankheitsprozesse: Schwerpunkt - Telomererhaltung
2. Integrative Multi-omische Charakterisierung: Schwerpunkt - Ultra-seltene Sarkome
3. Entwicklung von innovativen therapeutischen Ansätzen: Schwerpunkt - Gezielter Proteinabbau durch “molecular glues”
Diese Studien werden das Verständnis der Sarkomentstehung verbessern und Ziele für eine biologische Stratifizierung und eine auf molekulare Mechanismen gestützte therapeutische Intervention identifizieren.
Projekte
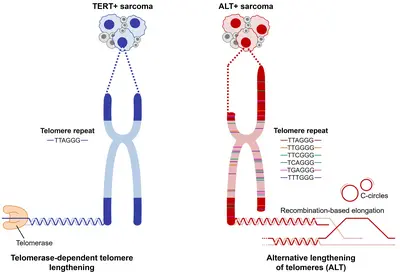
Telomere sind Nukleotidsequenzen, die aus 5'-TTAGGG-3'-Tandemwiederholungen bestehen und eine wichtige Rolle bei der Aufrechterhaltung der genomischen Integrität spielen, indem sie die Enden der Chromosomen mit Hilfe des Telomer-Shelterin-Komplexes vor DNA-Schäden schützen. Die Replikation der Telomere wird von der Telomerase durchgeführt. Um replikative Unsterblichkeit zu erreichen, reaktivieren etwa 85 % der Krebsarten die TERT-Expression. Die verbleibenden 15 % der Krebsarten erhalten die Telomerlänge durch einen telomeraseunabhängigen Mechanismus aufrecht, der als alternative Verlängerung der Telomere (ALT) bezeichnet wird. Unsere frühere Arbeit (Chudasama et al. Nat Comms 2018) und die anderer Forscher hat gezeigt, dass ALT ein häufiges Merkmal von Sarkomen ist, das zur Tumorprogression beiträgt. Da es sich bei ALT um einen krebszellspezifischen Prozess handelt, stellt sie ein attraktives therapeutisches Ziel dar, allerdings ist die Landschaft der auf ALT abzielenden Medikamente nicht gut etabliert. Die Untersuchung von Genen, die an der ALT beteiligt sind, könnte neue Biomarker für eine gezielte Behandlung dieser aggressiven Tumoren aufzeigen.
Bisher haben wir >800 menschliche Sarkom-Tumorproben untersucht, um die Häufigkeit von ALT in verschiedenen Sarkom-Untergruppen zu ermitteln. Nach dieser Stratifizierung haben wir eine integrative Multi-omics-Analyse durchgeführt, um genetische Veränderungen zu identifizieren, die ALT-positive Tumoren von ALT-negativen Tumoren unterscheiden, um ALT-assoziierte Aberrationen zu ermitteln, die direkt oder über sekundäre Abhängigkeiten angegriffen werden können. Anhand eines großen Panels seltener Sarkom-Zelllinien und von Patienten stammenden Xenotransplantaten validieren wir die „Medikamentenfähigkeit“ der in Frage kommenden Veränderungen und charakterisieren diese mechanistisch mit Hilfe der Phospho-Proteomik, um die Grundlage für die Auswahl ALT-spezifischer Ziele für die klinische Bewertung zu schaffen. Darüber hinaus entwickeln wir Methoden, um die Auswirkungen der Heterogenität der Telomererhaltungsmechanismen mithilfe von maschinellen Lernwerkzeugen (Belova et al. 2023 biorXiv) und räumlich aufgelösten Technologien (Frank et al. Nucleic Acids Res 2022) zu untersuchen.
Die begrenzte Verfügbarkeit von Tumorproben, das Fehlen von Modellsystemen und die geringere Anzahl von Patienten für das Verständnis der Krankheitsbiologie und die Entwicklung von Arzneimitteln sind kritische Hürden für das Vorantreiben der Grundlagen-, translationalen und klinischen Forschung bei seltenen Krebsarten (<6 Fälle pro 100.000/Jahr) wie dem Sarkom. Eine Untergruppe von Sarkomen, die so genannten ultra-seltenen Sarkome (≤1 Fall pro 1.000.000, Stacchiotti S et al, 2021, Cancer, 27:2934), sind äußerst selten, was die oben genannten Herausforderungen noch erheblich verschärft. Unser Team möchte die Erforschung ultra-seltener Sarkome vorantreiben, indem es eine eingehende molekulare Charakterisierung von Tumorproben durchführt, um die Diagnose, Klassifizierung, Identifizierung pathognomonischer Veränderungen, Modellentwicklung und Ansatzpunkte für eine biologisch gesteuerte Behandlung zu ermöglichen.
Follicular Dendritic Cell Sarcoma
One example of ultra-rare sarcoma is follicular dendritic sarcoma (FDCS) that originates from follicular dendritic cells (FDCs). Diagnosis of FDCS is difficult due to histological similarities with many epithelial, mesenchymal, meningeal, or lymphoid malignancies. FDCS patients have poor prognosis and are still commonly treated with a lymphoma chemotherapy regimen (CHOP) despite evidence of mesenchymal origin of FDCs. We have assembled a cohort of >30 FDCS samples and have characterized the genomic and transcriptomic landscape of alterations, which led to identification of previously not described targetable lesions and oncogenic mechanisms, such as BRAFV600E mutations and telomerase overexpression by enhancer hijacking. Results of our analysis have led to clinical benefit to FDCS patient and we have identified a rare histological subtype of this ultra-rare sarcoma. We continue to characterize this intriguing disease using DNA methylation landscape and spatial technologies.
Alveoläres Weichteilsarkom
Das alveoläre Weichteilsarkom (ASPS) ist ein weiteres sehr seltenes Sarkom mit schlechter Prognose im metastatischen Stadium. ASPS wird durch eine (X;17) (p11;q25)-Translokation zur Bildung des ASPSCR1-TFE3-Fusionsonkoproteins angetrieben, wobei nur wenige zusätzliche genomische Veränderungen wie Mutationen oder strukturelle Varianten auftreten. Wir waren fasziniert von der Feststellung, dass ASPS-Tumoren eine starke Infiltration von Immunzellen aufweisen, was nach dem derzeitigen Dogma kein häufiges Merkmal von Tumoren mit „stummen“ Genomen ist. Darüber hinaus zeigen bestimmte Patienten mit ASPS-Tumoren ein außergewöhnliches Ansprechen auf Checkpoint-Inhibition, was jedoch nicht mit der Expressionshöhe von Immun-Checkpoint-Molekülen korreliert. Wir setzen Transkriptom-basierte Immunprofilierungsansätze und räumliche Proteomik ein, um 1) die Geheimnisse der Chemotaxis von Immunzellen zu lüften, 2) einen Biomarker für das Ansprechen auf eine Checkpoint-Blockade-Behandlung zu identifizieren und 3) neue Mechanismen und Schwachstellen der fusionsbedingten Modulation der immunologischen Mikroumgebung zu erkennen. Zu diesem Zweck haben wir in Zusammenarbeit mit der Gruppe von Ana Banito (Weichteilsarkome, DKFZ und KiTZ) ein immunkompetentes ASPS-Mausmodell entwickelt, das die präklinische Validierung von Targets und Biomarkern ermöglichen wird.
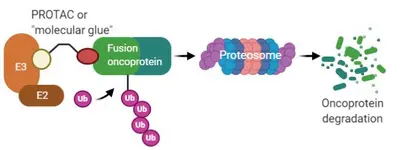
Ein Drittel der Sarkome ist durch wiederkehrende genetische Veränderungen, so genannte chromosomale Translokationen, gekennzeichnet, die durch Bruchstellen innerhalb zweier Zellgene entstehen und zur Bildung eines chimären Fusionsgens führen. In den meisten Fällen handelt es sich bei den Fusionsgenen in Sarkomen um Chromatinumwandlungsfaktoren oder Transkriptionsfaktoren. Die Genfusion führt zu einer Veränderung des Regulierungsmusters der Genexpression, was in den meisten Fällen zu einer verstärkten Proliferation, Resistenz gegen Apoptose, verstärkter Migration und Invasion führt. Da die Fusionsgene die onkogenen Triebkräfte sind, die nur in Tumorzellen exprimiert werden, stellen sie attraktive molekulare Ziele dar. Von Transkriptionsfaktoren abgeleitete Fusionsgene weisen jedoch keine enzymatische Aktivität auf und lassen sich daher nicht mit kleinen Molekülen hemmen. Ein vielversprechender Ansatz sind kleine Moleküle, die die Nähe zwischen E3-Ubiquitin-Ligasen und onkogenen Substraten herstellen und so die Substrate für den Abbau durch das Ubiquitin-Proteasom-System (UPS) anvisieren. Ein Beispiel für den Erfolg dieses Ansatzes sind Verbindungen wie Lenalidomid-Analoga, die auch als immunmodulatorische Medikamente (IMiDs) und Proteolyse-Zielchimären (PROTACs) bekannt sind. Mit Hilfe von Reporter-basierten funktionellen Genom-Screens, die mit Proteinbiochemie und strukturbiologischen Ansätzen gekoppelt sind, untersuchen wir die Proteinabbauwege von Sarkom-Fusionsgenen, um neue E3-Ligase-Fusions-Onkoprotein-Paare und Strukturen für „molekulare Kleber“ oder PROTAC-Designs zu nominieren.
Team
-

Dr. Priya Chudasama
Group leader
-

Wenxin Chao
MD student
-

Sherine Joanna Fredrick
Research Assistant
-

Hanna Frieß
Internship Student, Molecular Biosciences
Ausgewählte Publikationen
Belova T, Biondi N, Hsieh PH, Chudasama P, Kuijjer M.
ALT-FISH quantifies alternative lengthening of telomeres activity by imaging of single-stranded repeats
Frank L, Rademacher A,…, Fröhling S, Chudasama P, Rippe K.
Small molecule-induced polymerization triggers degradation of BCL6
Stabicki M, Yoon H, Koeppel J, …, Chudasama P, …, Ebert B.
Integrative genomic and transcriptomic analysis of leiomyosarcoma
Chudasama P., Mughal S.S et.al
Kontaktieren Sie uns