Molekulare Grundlagen thorakaler Tumoren
- Funktionelle und Strukturelle Genomforschung

Prof. Dr. Rocio Sotillo Roman
Group Leader
Unsere Forschung konzentriert sich darauf, die genetische und molekulare Grundlage von Lungen- und Brustkrebs zu verstehen, indem wir verschiedene onkogene Treiber und spezifische genetische Veränderungen untersuchen, die die Krebsprogression vorantreiben. Mit Hilfe von fortschrittlichen genetisch veränderten Mausmodellen untersuchen wir, wie unterschiedliche Genexpressionsprofile die Aggressivität von Tumoren und die Ansprechbarkeit auf Behandlungen beeinflussen.
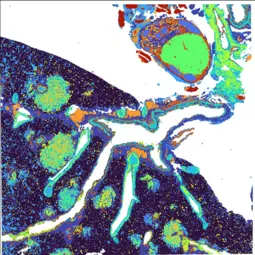
Unsere Forschung

Unsere Forschung konzentriert sich auf das Verständnis der genetischen und molekularen Grundlagen von Lungen- und Brustkrebs durch die Untersuchung verschiedener onkogener Triebkräfte und spezifischer genetischer Veränderungen, die das Fortschreiten des Krebses fördern. Mit Hilfe fortschrittlicher gentechnisch veränderter Mausmodelle untersuchen wir, wie unterschiedliche Genexpressionsprofile die Aggressivität des Tumors und das Ansprechen auf eine Behandlung beeinflussen. Ein wichtiger Teil unserer Forschung besteht in der Identifizierung von Biomarkern, die schlechte therapeutische Ergebnisse vorhersagen, mit dem Ziel, präzisere Krebstherapien zu entwickeln.
Methoden und Technologien: Unsere Forschung setzt mehrere Spitzentechnologien ein:
- Gentechnisch veränderte Mausmodelle zur genauen Nachahmung von menschlichem Krebs.
- CRISPR/Cas9-Technologie für präzise genetische Veränderungen zur Untersuchung der Auswirkungen bestimmter Gene auf Krebs.
- Lineage Tracing: Diese Technik verfolgt die Entwicklung und Differenzierung von Krebszellen und bietet Einblicke in die Tumorentwicklung.
- Epigenom- und Einzelzell-Transkriptomanalysen helfen uns, die Genexpression und regulatorische Veränderungen auf Einzelzellebene zu untersuchen, was unser Verständnis des Verhaltens von Krebszellen verbessert.
- 3D-In-vitro-Kultursysteme, die es uns ermöglichen, das Krebswachstum in einer Umgebung zu untersuchen, die den physiologischen Bedingungen sehr nahe kommt.
Ziele und gesellschaftliche Relevanz
Das übergreifende Ziel unserer Forschung ist es, das Verständnis der Krebsbiologie zu verbessern, um neue therapeutische Ziele und Strategien zu entdecken. Indem wir die molekularen Mechanismen der Tumorprogression und der genetischen Instabilität entschlüsseln, wollen wir wirksamere Behandlungen entwickeln. Unsere Bemühungen unterstützen das Deutsche Krebsforschungszentrum bei der Bewältigung der großen Herausforderungen in der Krebsforschung, insbesondere bei der Identifizierung und Überwindung von Resistenzmechanismen, um die Ergebnisse für die Patienten zu verbessern.
Projekte
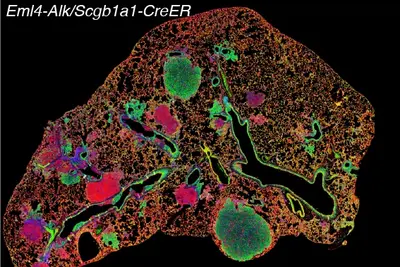
Entschlüsselung der genetischen Hintergrund des nicht-kleinzelligen Lungenkrebses: Fusionsvarianten und therapeutische Möglichkeiten
Nicht-kleinzelliger Lungenkrebs (non-small cell lung cancer, NSCLC) ist eine der häufigsten und tödlichsten Krebsarten weltweit. Eine wichtige Ursache für NSCLC ist die Fusion zwischen den Genen anaplastische Lymphomkinase (ALK) und echinoderm microtubule-associated protein-like 4 (EML4). Bislang wurden etwa 15 verschiedene EML4-ALK-Fusionsvarianten identifiziert. Diese weisen jeweils unterschiedliche Merkmale auf, die sich auf ihre Stabilität, ihre genomische Lokalisierung, ihr Ansprechen auf zielgerichtete Therapien und ihre Auswirkungen auf die Prognose auswirken.
Unsere Forschung ist darauf ausgerichtet, die komplizierten Mechanismen zu entschlüsseln, die die Entwicklung und das Fortschreiten dieser Fusionsvarianten steuern. Wir sind besonders daran interessiert, wie gleichzeitig auftretende genetische Veränderungen, wie beispielsweise der Verlust von TP53, das Verhalten dieser Varianten beeinflussen und neue therapeutische Ziele identifizieren. Neben EML4-ALK-Fusionen erforscht unser Team auch andere chromosomale Rearrangements, die zur Pathogenese des NSCLC beitragen, einschließlich der Auswirkungen des Verlusts des Y-Chromosoms und der Rolle spezifischer epigenetischer Regulatoren wie Kdm5d und Uty bei dieser Tumorart.
Um diese genetischen Feinheiten zu untersuchen, nutzen wir CRISPR/Cas9 Gene Editing, um gentechnisch veränderte Mausmodelle zu erzeugen. Diese ermöglichen die induzierbare Expression spezifischer Genfusionen und die gezielte Inaktivierung von Genen, die mit NSCLC in Verbindung stehen. Unser Ansatz umfasst modernste Techniken, darunter speziell angepasste in-vivo-Tumormodelle, die die menschliche Tumorerkrankungen nachahmen, Organoide aus Maus- und Patientengewebe sowie sequenzielle Untersuchungen von Genveränerderungen in vivo. Diese Methodiken bildeen nicht nur die histopathologischen und molekularen Merkmale des menschlichen Lungenkrebses nach, sondern bietet auch eine herausragende Plattform für die Entschlüsselung der molekularen Treiber des NSCLC und die Entwicklung bahnbrechender Behandlungsmöglichkeiten.
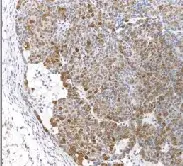
Erforschung der evolutionäre Dynamik von Her2+ Brustkrebs
Her2+ Brustkrebs ist für seine dynamische Eigenschaften bekannt, die sich als Reaktion auf Therapien und Immuntherapien ständig weiterentwickelt und anpasst. Unsere Forschung konzentriert sich auf das Verständnis der molekularen und genetischen Mechanismen, die diese Entwicklung vorantreiben. Wir verwenden komplexe Mausmodelle, um neue Schwachstellen zu erkennen und den therapeutisch Erfolg zu verbessern.
Unsere bisherigen Arbeiten haben Aufschluss darüber gegeben, wie die chromosomale Instabilität (chromosomal instability, CIN) zum Fortschreiten von Brustkrebs beiträgt. Wir haben endeckt, dass die Überexpression von PLK1 die Immunsuppression durch Seneszenz-assoziierte sekretorische Phänotypen (SASPs) und NF-κβ-Signalgebung fördert. Darüber hinaus hat unsere Forschung gezeigt, dass FOXM1 aneuploiden Tumorzellen bei der Bewältigung von mitotischem Stress hilft, was es zu einem vielversprechenden therapeutischen Ziel macht.
In jüngster Zeit hat sich unser Schwerpunkt auf APOBEC3B (A3B) verlagert, ein DNA-editierendes Enzym, das Mutationen einführt und damit die Tumorevolution vorantreibt. Mit Hilfe eines Her2-A3B-induzierbaren Mausmodells wollen wir herausfinden, wie A3B die Entstehung und das Fortschreiten von Her2+ Brustkrebs beeinflusst.
Durch die Integration von Studien zur chromosomalen Instabilität, zu Mutationsprozessen und zur Mikroumgebung des Tumors setzen wir das komplexe Puzzle der Brustkrebsevolution zusammen. Unser umfassender Ansatz, bei dem wir verschiedene Mausmodelle verwenden, führt uns zu wirksameren Therapiestrategien, die Hoffnung zur Bekämpfung diese hochgradig adaptive Erkrankung bietet.
Team
- Profil anzeigen

Prof. Dr. Rocio Sotillo Roman
Group Leader
- Profil anzeigen

Maria Bandeira Ferreira Ramos
PhD student
- Profil anzeigen

Maria Capone
PhD student
- Profil anzeigen

Maria Teresa Castillo Alvarez
PhD student
-

Hilary Ann Davies-Rück
Administrative assistant
- Profil anzeigen

Can Gürkaslar
Lab technician
-
Janina Hattemer
Visiting student
- Profil anzeigen

Luisa Kinas
Master student
- Profil anzeigen

Amelie Mahr
PhD student
- Profil anzeigen

Mulham Najajreh
PhD student
- Profil anzeigen

Lukas Otto
Master student
- Profil anzeigen

Francisco Javier Rios Sola
Erasmus Student
- Profil anzeigen

Dr. Kalman Somogyi
Senior scientist
Publikationen
Monfort-Vengut A, Sanz-Gómez N, Ballesteros-Sánchez S, Ortigosa B, Cambón A, Ramos M, Lorenzo ÁM, Escribano-Cebrián M, Rosa-Rosa JM, Martínez-López J, Sánchez-Prieto R, Sotillo R, de Cárcer G.
de Jaime-Soguero A, Hattemer J, Bufe A, Haas A, van den Berg J, van Batenburg V, Das B, di Marco B, Androulaki S, Böhly N, Landry JJM, Schoell B, Rosa VS, Villacorta L, Baskan Y, Trapp M, Benes V, Chabes A, Shahbazi M, Jauch A, Engel U, Patrizi A, Sotillo R, van Oudenaarden A, Bageritz J, Alfonso J, Bastians H, Acebrón SP.
Chronic spindle assembly checkpoint activation causes myelosuppression and gastrointestinal atrophy.
Karbon G, Schuler F, Braun VZ, Eichin F, Haschka M, Drach M, Sotillo R, Geley S, Spierings DC, Tijhuis AE, Foijer F, Villunger A.
Horvat NK, Chocarro S, Marques O, Bauer TA, Qiu R, Diaz-Jimenez A, Helm B, Chen Y, Sawall S, Sparla R, Su L, Klingmüller U, Barz M, Hentze MW, Sotillo R, Muckenthaler MU.
Diaz-Jimenez A, Ramos M, Helm B, Chocarro S, Frey DL, Agrawal S, Somogyi K, Klingmüller U, Lu J, Sotillo R.
Cai L, Gao Y, DeBerardinis RJ, Acquaah-Mensah G, Aidinis V, Beane JE, Biswal S, Chen T, Concepcion-Crisol CP, Grüner BM, Jia D, Jones R, Kurie JM, Lee MG, Lindahl P, Lissanu Y, Lorz Lopez MC, Martinelli R, Mazur PK, Mazzilli SA, Mii S, Moll H, Moorehead R, Morrisey EE, Ng SR, Oser MG, Pandiri AR, Powell CA, Ramadori G, Santos Lafuente M, Snyder E, Sotillo R, Su KY, Taki T, Taparra K, Xia Y, van Veen E, Winslow MM, Xiao G, Rudin CM, Oliver TG, Xie Y, Minna JD.
Alonso de la Vega A, Temiz NA, Tasakis R, Somogyi K, Salgueiro L, Zimmer E, Ramos M, Diaz-Jimenez A, Chocarro S, Fernández-Vaquero M, Stefanovska B, Reuveni E, Ben-David U, Stenzinger A, Poth T, Heikenwälder M, Papavasiliou N, Harris RS, Sotillo R.
Kandala S, Ramos M, Voith von Voithenberg L, Diaz-Jimenez A, Chocarro S, Keding J, Brors B, Imbusch CD, Sotillo R.
Durfee C, Temiz NA, Levin-Klein R, Argyris PP, Alsøe L, Carracedo S, Alonso de la Vega A, Proehl J, Holzhauer AM, Seeman ZJ, Liu X, Lin YT, Vogel RI, Sotillo R, Nilsen H, Harris RS.
Raach B, Bundgaard N, Haase MJ, Starruß J, Sotillo R, Stanifer ML, Graw F.
Pan F, Chocarro S, Ramos M, Chen Y, Alonso de la Vega A, Somogyi K, Sotillo R.
Durfee C, Temiz NA, Levin-Klein R, Argyris PP, Alsøe L, Carracedo S, de la Vega AA, Proehl J, Holzhauer AM, Seeman ZJ, Lin YT, Vogel RI, Sotillo R, Nilsen H, Harris RS.
Chen Y, Toth R, Chocarro S, Weichenhan D, Hey J, Lutsik P, Sawall S, Stathopoulos GT, Plass C, Sotillo R.
Salgueiro L, Kummer S, Sonntag-Buck V, Weiß A, Schneider MA, Kräusslich HG, Sotillo R.
Lamort AS, Kaiser JC, Pepe MAA, Lilis I, Ntaliarda G, Somogyi K, Spella M, Behrend SJ, Giotopoulou GA, Kujawa W, Lindner M, Koch I, Hatz RA, Behr J, Sotillo R, Schamberger AC, Stathopoulos GT.
Alikhanyan K, Chen Y, Somogyi K, Kraut S, Sotillo R.
Bauer TA, Horvat NK, Marques O, Chocarro S, Mertens C, Colucci S, Schmitt S, Carrella LM, Morsbach S, Koynov K, Fenaroli F, Blümler P, Jung M, Sotillo R, Hentze MW, Muckenthaler MU, Barz M.
Rubio T, Weyershaeuser J, Montero MG, Hoffmann A, Lujan P, Jechlinger M, Sotillo R, Köhn M.
Mayakonda A, Schönung M, Hey J, Batra RN, Feuerstein-Akgoz C, Köhler K, Lipka DB, Sotillo R, Plass C, Lutsik P, Toth R.
Alikhanyan K, Chen Y, Kraut S, Sotillo R.
Salgueiro L, Buccitelli C, Rowald K, Somogyi K, Kandala S, Korbel JO, Sotillo R.
Raimondi F, Inoue A, Kadji FMN, Shuai N, Gonzalez JC, Singh G, de la Vega AA, Sotillo R, Fischer B, Aoki J, Gutkind JS, Russell RB.
Spella M, Lilis I, Pepe MA, Chen Y, Armaka M, Lamort AS, Zazara DE, Roumelioti F, Vreka M, Kanellakis NI, Wagner DE, Giannou AD, Armenis V, Arendt KA, Klotz LV, Toumpanakis D, Karavana V, Zakynthinos SG, Giopanou I, Marazioti A, Aidinis V, Sotillo R, Stathopoulos GT.
Christopoulos P, Kirchner M, Bozorgmehr F, Endris V, Elsayed M, Budczies J, Ristau J, Penzel R, Herth FJ, Heussel CP, Eichhorn M, Muley T, Meister M, Fischer JR, Rieken S, Lasitschka F, Bischoff H, Sotillo R, Schirmacher P, Thomas M, Stenzinger A.
de Cárcer G, Venkateswaran SV, Salgueiro L, El Bakkali A, Somogyi K, Rowald K, Montañés P, Sanclemente M, Escobar B, de Martino A, McGranahan N, Malumbres M, Sotillo R.
Havas KM, Milchevskaya V, Radic K, Alladin A, Kafkia E, Garcia M, Stolte J, Klaus B, Rotmensz N, Gibson TJ, Burwinkel B, Schneeweiss A, Pruneri G, Patil KR, Sotillo R, Jechlinger M.
Domingues PH, Nanduri LSY, Seget K, Venkateswaran SV, Agorku D, Viganó C, von Schubert C, Nigg EA, Swanton C, Sotillo R, Bosio A, Storchová Z, Hardt O.
Buccitelli C, Salgueiro L, Rowald K, Sotillo R, Mardin BR, Korbel JO.
Weischenfeldt J, Dubash T, Drainas AP, Mardin BR, Chen Y, Stütz AM, Waszak SM, Bosco G, Halvorsen AR, Raeder B, Efthymiopoulos T, Erkek S, Siegl C, Brenner H, Brustugun OT, Dieter SM, Northcott PA, Petersen I, Pfister SM, Schneider M, Solberg SK, Thunissen E, Weichert W, Zichner T, Thomas R, Peifer M, Helland A, Ball CR, Jechlinger M, Sotillo R, Glimm H, Korbel JO.
Rowald K, Mantovan M, Passos J, Buccitelli C, Mardin BR, Korbel JO, Jechlinger M, Sotillo R.
Schvartzman JM, Duijf PH, Sotillo R, Coker C, Benezra R.
Aneiros-Fernandez J, Arias-Santiago S, Sotillo R, Menjon-Beltran S, Concha A.
Sotillo R, Schvartzman JM, Socci ND, Benezra R.
Schvartzman JM, Sotillo R, Benezra R.
Sotillo R, Schvartzman JM, Benezra R.
Diaz-Rodríguez E, Sotillo R, Schvartzman JM, Benezra R.
Niault T, Hached K, Sotillo R, Sorger PK, Maro B, Benezra R, Wassmann K.
Sotillo R, Hernando E, Díaz-Rodríguez E, Teruya-Feldstein J, Cordón-Cardo C, Lowe SW, Benezra R.
Barbacid M, Ortega S, Sotillo R, Odajima J, Martín A, Santamaría D, Dubus P, Malumbres M.
Sotillo R, Renner O, Dubus P, Ruiz-Cabello J, Martín-Caballero J, Barbacid M, Carnero A, Malumbres M.
Malumbres M, Sotillo R, Santamaría D, Galán J, Cerezo A, Ortega S, Dubus P, Barbacid M.
Ortega S, Prieto I, Odajima J, Martín A, Dubus P, Sotillo R, Barbero JL, Malumbres M, Barbacid M.
Martín J, Hunt SL, Dubus P, Sotillo R, Néhmé-Pélluard F, Magnuson MA, Parlow AF, Malumbres M, Ortega S, Barbacid M.
Malumbres M, Hunt SL, Sotillo R, Martín J, Odajima J, Martín A, Dubus P, Ortega S, Barbacid M.
Sotillo R, Dubus P, Martín J, de la Cueva E, Ortega S, Malumbres M, Barbacid M.
Sotillo R, García JF, Ortega S, Martin J, Dubus P, Barbacid M, Malumbres M.
Latres E, Malumbres M, Sotillo R, Martín J, Ortega S, Martín-Caballero J, Flores JM, Cordón-Cardo C, Barbacid M.
Ausgewählte Publikationen
Diaz-Jimenez, A., Shuldiner, E. G., Somogyi, K., Gonzalez, O., Akkas, F., Murray, C. W., Andrejka, L., Tsai, M. K., Brors, B., Sivakumar, S., Sisoudiya, S. D., Sokol, E. S., Petrov, D. A., Winslow, M. M., & Sotillo, R.
Diaz-Jimenez A, Ramos M, Helm B, Chocarro S, Frey DL, Agrawal S, Somogyi K, Klingmüller U, Lu J, Sotillo R.
Alonso de la Vega A, Temiz NA, Tasakis R, Somogyi K, Salgueiro L, Zimmer E, Ramos M, Diaz-Jimenez A, Chocarro S, Fernández-Vaquero M, Stefanovska B, Reuveni E, Ben-David U, Stenzinger A, Poth T, Heikenwälder M, Papavasiliou N, Harris RS, Sotillo R
Kontaktieren Sie uns


