Funktionelle und Molekulare Emissions-Computertomographie
Funktionelle und molekulare Emissions-Computertomographie

Unsere Forschungsgruppe ist darauf ausgerichtet, den diagnostischen Wert von emissions-tomographischen Bildgebungssystemen zu verbessern und zu steigern, insbesondere im Hinblick auf Krebs. Dazu gehören die nuklearmedizinischen Bildgebungsverfahren Positronen-Emissions-Tomographie (PET) und Einzelphotonen-Emissions-Computertomographie (SPECT) sowohl für klinische als auch für präklinische Anwendungen. Mit Schwerpunkt auf der präklinischen Bildgebung bei Kleintieren gehören dazu auch die optischen Modalitäten Biolumineszenz-Bildgebung (BLI) sowie fluoreszenzvermittelte Bildgebung (FMI) und fluoreszenzdiffusionsoptische Tomographie (DOT).
Forschungsthemen
- Multimodale Emissionsbildgebungssysteme mit FMT, BLI, CT, SPECT
- Synchromodale Emissionsabbildungssysteme mit FMT, BLI, MRI, PET
- Simulationswerkzeuge für multimodale Bildgebungssysteme mit keV- und eV-Photonen
- Monolithische PET-Detektoreinheiten für präklinische und Ganzkörper-PET
- Plenoptische Bildgebungssysteme für in vivo FMT, BLI, DOT
- Anthropomorphe und zoomorphe Phantome mit integrierten Tumorgeweben
Unsere Arbeitsgruppe ist Mitglied in der Crystal Clear Collaboration.
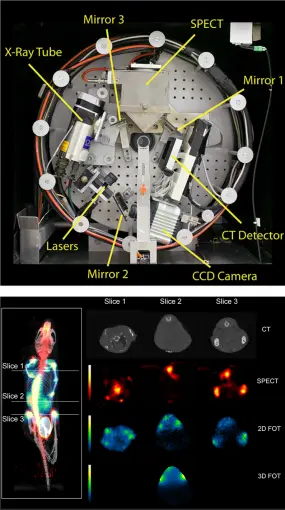
Einige unserer Projekte werden in einem echten multimodalen Instrumentenkontext geplant und umgesetzt. Dazu gehören Forschungsarbeiten zu PET-CT und PET-MRT (klinische Systeme) sowie zur optischen Bildgebung in Kombination mit anderen Modalitäten (großes Sichtfeld). Der weltweit erste dreifach modale SPECT-FMI/BLI-CT-Maus-Imager (linke Abbildung, oben: Frontansicht des trimodalen Bildgebungssystems, unten: fusionierte MIP-Ansicht der rekonstruierten CT-, SPECT- und 2D-FMT-Daten (Referenz)) wurde in dieser Forschungsgruppe gebaut.
Das Ziel dieses Instruments war die gleichzeitige Detektion von radioaktiv markierten pharmazeutischen Verteilungen (SPECT), fluoreszierenden molekularen Markern im nahen Infrarot (fluoreszenzvermittelte Bildgebung und Tomographie (FMI/FMT)) und/oder Biolumineszenz-Bildgebung (BLI) und hochauflösender Röntgentomographie (CT) mit axial unverschobenen (d.h. identischen), räumlich überlappenden Sichtfeldern (FOV) aller beteiligten Submodalitäten. Für die SPECT-Bildgebung wird ein kompakter Gamma-Detektor (Thomas Jefferson National Accelerator Facility, USA) eingesetzt. Er besteht aus einem 2x2-Array positionsempfindlicher Hamamatsu H8500-Photomultiplier-Röhren, die mit einem 66x66-Array optoentkoppelter 1,3x1,3x6 mm3 NaI(Tl)-Kristallelemente (St. Gobain) verbunden sind, was eine Gesamtdetektorfläche von 10x10 cm2 ergibt. An die Kamera können verschiedene Kollimatoren (Lochblende, Fächerstrahl, Parallelstrahl) für spezifische Abbildungszwecke angebracht werden. Als optisches Detektor-Teilsystem wird eine hochauflösende gekühlte CCD-Kamera der ORCA AG (Hamamatsu) verwendet, die einen progressiv abtastenden Interline-CCD-Chip mit einem 1344x1024-Pixel-Array und 12-Bit-Digitalausgang enthält. An der Gantry können verschiedene Laserquellen, die je nach Wellenlänge und Lichtleistung ausgewählt werden können, montiert werden. Das Röntgen-CT-Subsystem verwendet eine Apogee-Röntgenröhre der Serie 5000 (Oxford Instruments) mit einer maximalen Leistung von 50 W, bei 4 bis 50 kV, 0 bis 1 mA. Der Brennfleck hat eine Größe von 35µm und der Kegelwinkel beträgt 24 Grad. Der Röntgendetektor ist eine Shad-o-Box 2048 (Rad-icon Imaging Corp.), die einen 50x100 mm2 großen Gd2O2S-Szintillatorschirm enthält, der in direktem Kontakt mit einem CMOS-Fotodiodenarray mit 48µm Sensorpixelgröße steht. Das Integrationskonzept ist vollständig modular aufgebaut, wobei alle Komponenten auf einem gemeinsamen Gantry-System montiert sind, so dass eine Vielzahl von Anwendungen durchgeführt werden kann. Dieses Design ermöglicht eine höchstmögliche sub- und/oder multi-modale Leistung. Die gesamte Baugruppe ist in einem lichtdichten und gegen Gammastrahlen abgeschirmten Gehäuse untergebracht, das auf einem beweglichen Wagen montiert ist, der ein weiteres Gehäuse enthält, in dem sich die gesamte Elektronik für die Kamerasteuerung, die Datenauslesung, das Laserlicht, die Gantry- und Lineartischbewegung sowie die Hochspannungs- und Stromversorgung und die Arbeitsplatzrechner befinden. Mit unserem trimodalen Imager ist eine einheitliche simultane Datenerfassung, Bildrekonstruktion und fusionierte planare und tomographische Bilddarstellung möglich, so dass inhärent registrierte, potenziell dreidimensionale Fluoreszenz- und Radiopharmaka-Verteilungskarten mit molekularen und funktionellen Informationen erstellt werden können, die mit der Anatomie des abgebildeten Objekts korreliert sind, das durch Röntgenstrahlen dargestellt wird.
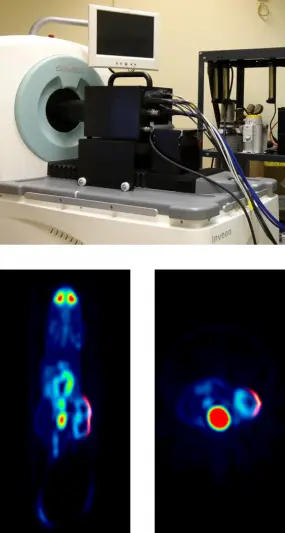
Um das zu ermöglichen, was wir synchromodale optische Bildgebung nennen, haben wir die erste plenoptische Kamera für die diagnostische Forschung erfunden, die nicht nur dreidimensionale (Beleuchtungs- und Fluenz-) Projektionen des abgebildeten Objekts erzeugt, sondern auch mit PET und MRT integriert und verwendet werden kann (Referenz). Das weltweit erste simultan aufgenommene [18F]FDG PET - pVEGF Luc BLI plenoptische Bild ist in der linken Abbildung zu sehen (oben: experimentelles plenoptisches System, integriert in ein Siemens Inveon PET zur simultanen Datenerfassung, unten: fusionierte PET- und OI-Daten (Referenz)).
Es wird ein komplettes Instrumentarium und ein mathematischer Rahmen für die präklinische plenoptische Bildgebung (POI) entwickelt, bei der die optischen Daten mit Hilfe eines auf einem Mikrolinsen-Array (MLA) basierenden Lichtdetektors (MLA-D) erfasst werden. Der MLA-D wurde entwickelt, um einzigartige POI zu ermöglichen, insbesondere im synchronen Betrieb mit sekundären Bildgebungsmodalitäten wie PET oder MRI. Ein MLA-D besteht aus einem (großflächigen) Photonensensor-Array, einem angepassten MLA zur Definition des Sichtfelds und einer Septum-Maske mit spezifischer Geometrie aus eloxiertem Aluminium, die zwischen dem Sensor und dem MLA positioniert wird, um das Übersprechen des Lichts zu unterdrücken und das Hochfrequenz-Interferenzsignal des Sensors abzuschirmen (unerlässlich beim Einsatz in einem MRT-System). Das Software-Framework ist zwar für jeden MLA-D frei parametrierbar, aber auf ein POI-Prototypsystem für präklinische synchromodale Bildgebungsanwendungen zugeschnitten, das eine Vielzahl von zylindrisch montierten, in einer Gantry angebrachten, gleichzeitig arbeitenden MLA-Ds umfasst. Im synchromodalen Betrieb können die rekonstruierten tomographischen Volumendaten für die co-modale Bildfusion und auch als Grundlage für die Schätzung der 3D-Oberfläche des abgebildeten Objekts mit Hilfe des Gradientenvektorflusses verwendet werden. Überlagertes planares oder oberflächenorientiertes inverses Mapping kann durchgeführt werden, um die Emissionslichtkarte zu schätzen und mit der Grenze des abgebildeten Objekts zu verschmelzen. Durch Triangulation und anschließende optische Rekonstruktion kann die interne dreidimensionale Emissionslichtverteilung geschätzt werden. Der Rahmen ist anfällig für eine Reihe von Variablen, die die Konvergenz und die Berechnungsgeschwindigkeit steuern.

Eines unserer Schwerpunktthemen ist die Monte-Carlo-Simulation der Emissionsbildgebung, d. h. der Ausbreitung von eV- und keV-Photonen in heterogenen Phantomen und verschiedenen Scanner- und Detektorgeometrien, und zwar innerhalb eines einzigartigen, einfach zu bedienenden multimodalen Frameworks, Musiré, das nicht nur die Verwendung von 3D-Netzen und/oder voxelierten Daten für alle beteiligten Modalitäten mit nahtloser Transformation und Anpassung zwischen ihnen ermöglicht, sondern auch zum ersten Mal die Integration von Tumorläsionen, die durch (Tumor-)Zellwolken definiert sind, die das Ergebnis biologischer Krebssimulationen sind.
Mit Musiré wird ein softwarebasierter Arbeitsablauf zur Verwaltung der Ausführung von Simulationen und Bildrekonstruktionen für SPECT-, PET-, CBCT-, MRI-, BLI- und FMI-Pakete in einzelnen und multimodalen biomedizinischen Bildgebungsanwendungen vorgeschlagen. Der Arbeitsablauf besteht aus einem Bash-Skript, dessen Zweck es ist, dem Benutzer eine Schnittstelle zu bieten und den Datenfluss zwischen den einzelnen Programmen für die Simulation und Rekonstruktion zu organisieren. Zu den derzeit integrierten Simulationsprogrammen gehören GATE für die Monte-Carlo-Simulation von SPECT, PET und CBCT, SpinScenario für die Simulation von MRI und Lipros für die Monte-Carlo-Simulation von BLI und FMI. Zu den derzeit integrierten Bildrekonstruktionsprogrammen gehören CASToR für SPECT und PET sowie RTK für CBCT.
Der MetaImage (mhd)-Standard wird für voxelisierte Phantom- und Bilddatenformate verwendet. Meshlab-Projekt-Container (mlp) mit Polygonnetzen und Punktwolken, die durch das Stanford-Dreiecksformat (ply) definiert sind, werden zur Darstellung anatomischer Strukturen für optische Simulationen und zur Darstellung von Tumorzellinserts verwendet. Eine Reihe von Hilfsprogrammen wurde für die Datentransformation und die adaptive Parameterzuweisung entwickelt. Der Software-Workflow nutzt die vollautomatische Verteilung auf und Konsolidierung von einer beliebigen Anzahl von Linux-Workstations und CPU-Kernen.
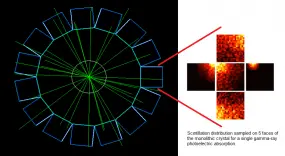
Mit diesem Projekt soll ein Beitrag zur Weiterentwicklung der PET-Instrumente und zur Entwicklung neuer multimodaler Techniken geleistet werden. Von präklinischen Systemen bis hin zu klinischen Ganzkörpersystemen liegt der Schwerpunkt auf der Untersuchung des Designs monolithischer Detektoren, vor allem durch den Einsatz von Computersimulationen und mit Unterstützung grundlegender experimenteller Tests. Der Simulationsansatz umfasst die vollständige Simulation der Szintillationsphotonen in den Kristallen und stellt daher ein leistungsfähiges Werkzeug dar, das zur Optimierung des ko-abhängigen geometrischen Scanner-Designs und des angewandten Algorithmus zur Bestimmung des Gammastrahlen-Interaktionspunkts eingesetzt wird. Ein Prototyp für einen multimodalen präklinischen Scanner, der die PET-Bildgebung mit der optischen Tomographie verbindet, wird derzeit mit Hilfe dieser Werkzeuge entwickelt.

Entwurf eines Tomographiesystems
Berührungslose optische In-vivo-Bildgebungssysteme, die speziell für die molekulare Biolumineszenz- und Fluoreszenzbildgebung (BLI, FMI) bei Kleintieren entwickelt wurden und linsenbasierte Kameras verwenden, sind in präklinischen Labors mittlerweile Standard. Solche Instrumente umfassen planare Bildgebungssysteme, die zweidimensionale (2D) Bilder eines dreidimensionalen (3D) In-vivo-Emissionsflusses aufnehmen. Bei der Durchführung planarer Lichtemissionsaufnahmen auf komplexen Oberflächengeometrien variieren die lokalen Emissionsstrahlintensitäten sowie deren räumliche Verteilung des induzierten Emissionsflusses in Abhängigkeit von Änderungen der Kameraposition und des Winkels in Bezug auf die Oberfläche des Tieres. Während entsprechende Messschwankungen durch die Erfassung von Lichtstrahlen orthogonal zu Oberflächenpunkten minimiert werden könnten, ist dies mit einer singulären Ganzkörper-2D-Lichtprojektion unmöglich zu erreichen.
Zur Lösung des Problems der In-vivo-Messung des Emissionslichtstroms, der aus der Hülle eines 3D-Objekts austritt, bei gleichzeitiger Erhaltung und Schätzung des a priori unbekannten 3D-Grenzraums, berichten wir über die Entwicklung und erste Simulationsergebnisse eines plenoptischen Bildgebungssystems, das speziell für In-vivo-BLI- und FMI-Anwendungen konzipiert wurde. Dank einer Vielzahl beliebig positionierbarer integrierter Laserdioden-Lichtquellen, die sowohl Punkt- als auch Hellfeldbeleuchtungen ermöglichen, ist das System in der Lage, multispektrale fluoreszenzvermittelte Tomographie (FMT) und diffusionsoptische Tomographie (DOT) durchzuführen, ohne dass ein zusätzliches Bildgebungsverfahren zur Oberflächendetektion erforderlich ist.
Es wurde ein plenoptisches Bildgebungssystem für präklinische In-vivo-Bildgebungsanwendungen entwickelt (Abbildung links). Bei der Aufnahme eines dreidimensionalen (3D) Objekts aus sechs plenoptischen Kamerapositionen werden die entsprechenden plenoptischen Rohdaten unter Hellfeldbeleuchtung gezeigt. Aufnahmesystem und verwendete Daten aus einer Simulationsstudie: Bildgebendes System bestehend aus 6 plenoptischen Kameras mit empfindlichen Bereichen von jeweils 25mm× 50 mm mit nahezu orthographischen Projektionen; in den Zwischenräumen zwischen den Lichtdetektoren befinden sich (einziehbare) diffuse Faserlinienquellen mit Detektorlänge zur Objektbeleuchtung (oben). Das segmentierte Volumen eines rekonstruierten Röntgen-CT-Maus-Datensatzes wird als identifizierter Input verwendet (Mitte). Daraus resultierende Rohdaten der plenoptischen Kamera, wie sie aus 6 Projektionswinkeln um 360° erkannt wurden (unten).
Computermathematik
Bei der rechnergestützten Bildgebung gibt es mehrere Strategien. Hier wird die Oberflächenrekonstruktion aus plenoptischen Mehrseitenprojektionsbilddaten beschrieben (Referenz). Die Technik ist für die In-vivo-Bildgebung von Kleintieren, insbesondere von Nacktmäusen, geeignet und erfordert keinen zusätzlichen Bildgebungsschritt (z. B. mittels einer sekundären strukturellen Modalität) oder zusätzliche Hardware (z. B. Laserscanning-Ansätze). Jeder potenzielle Punkt innerhalb des Sichtfeldes (FOV) wird durch ein vorgeschlagenes Fotokonsistenzmaß bewertet, das die Lichtinformationen des Sensorbildes nutzt, wie sie von Elementarbildern (EI) bereitgestellt werden. Da die Überlagerung benachbarter EI's komplementäre Informationen für jeden Punkt innerhalb des FOV liefert, wird die dreidimensionale (3D) Oberfläche des abgebildeten Objekts durch eine auf Graphenschnitten basierende Methode durch globale Energieminimierung geschätzt. Die vorgeschlagene Oberflächenrekonstruktion wird an simulierten MLA-D-Daten evaluiert, die ein rekonstruiertes Maus-Datenvolumen enthalten, das mit einem Röntgen-CT aufgenommen wurde. Im Vergleich zu einer zuvor vorgestellten, auf Rückprojektion basierenden Oberflächenrekonstruktionsmethode führt die vorgeschlagene Technik zu einer deutlich geringeren Fehlerrate. Während die auf Rückprojektion basierende Methode nicht in der Lage ist, konkave Bereiche aufzulösen, ist dies mit dem neuen Ansatz möglich. Unsere Ergebnisse zeigen außerdem, dass die vorgeschlagene Methode eine hohe Genauigkeit bei einer geringen Anzahl von Projektionen erreicht.
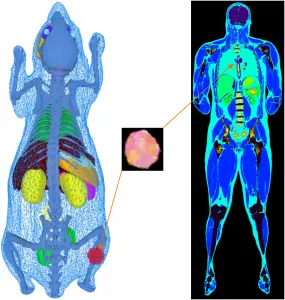
In unseren Simulationspaketen, wie z.B. Musiré (siehe auch Simulationswerkzeuge für die multimodale Bildgebung), kann eine einzige Objektbeschreibung (Phantom, Atlas) für die Simulation aller bildgebenden Modalitäten verwendet werden. Während für SPECT-, PET-, CT- und MRT-Simulationen in der Regel voxelierte Phantome verwendet werden, wird für die Simulation optischer Photonen (BLI, FMI) eine netzbasierte Darstellung anatomischer Strukturen bevorzugt, da die Begrenzung des abgebildeten Objekts entscheidende Informationen über den emittierten Photonenfluss liefert, die einen entscheidenden Faktor für die Genauigkeit der Bildrekonstruktion oder der inversen Lichtfeldabbildung darstellen.
Unsere Phantome umfassen (i) den dreidimensionalen (x, y, z) voxelisierten Gewebe-/Materialatlas, der als MetaIO (mhd) kodiert ist, und (ii) das dreidimensionale polygonale (Dreiecks-)Netz für die Geometrie des Gewebeatlasses, das die allgemeine Anatomie (und Objekte) darstellt und im Stanford-Dreiecksformat (ply) kodiert ist. Unsere Programme sind auch mit dreidimensionalen Punktwolken kompatibel, die im ply-Format kodiert sind. Eine Vielzahl von Netzen und Punktwolken kann in einem MeshLab-Projektcontainer (mlp) gekapselt werden. Daraus konvertieren unsere Frameworks die Objektbeschreibung automatisch in das passende Format für jeden Simulator. Der Mechanismus der Übergabe von Parameterdateien oder Skripten ermöglicht auch die Verwendung von simulatorspezifischen Phantomdarstellungsmodellen. Physikalische Attribute wie die Definition von Materialien, Dichten, Quellenaktivitäten, Farbstoffkonzentrationen, Relaxationszeiten und andere werden den mhd- oder ply-repräsentierten Atlasregionen durch einfache Look-up-Table-Dateien (dat) zugewiesen. Punktwolken sind für die Darstellung heterogener Tumorzellverteilungen (mit mehreren Läsionen) vorgesehen, wie sie von biologisch abgeleiteten Modellen für die Tumorwachstumsdynamik erzeugt werden. Anatomische Phantome können mit beliebig platzierbaren Tumorinserts erweitert werden. Diese Einsätze können beliebig feste, mehrläsige, nekrotische oder heterogene Gebilde mit Millionen von Zellen pro Kubikzentimeter Gewebe darstellen. Je nach der simulierten Bildgebungsmodalität können Punktwolken in ihrer nativen Auflösung verwendet werden. Dies wird für die optische Bildgebung eingesetzt, bei der die Biolumineszenz-Lichtverteilung direkt aus der räumlichen Tumorzellverteilung abgetastet wird. Da die Größe von Tumorzellen jedoch in der Größenordnung von 10 μm bis 50 μm liegt, wäre es ineffizient, diesen Grad an räumlicher Auflösung zu verwenden, z. B. bei der Simulation der nuklearen Bildgebung. Daher werden Punktwolken-Tumorinserts für andere Modalitäten automatisch auf die räumliche Auflösung des Wirtsphantoms heruntergerechnet. Durch die Verwendung spezifischer Befehlszeilenoptionen kann die Tumorzelldichte pro Voxel in einen Quellaktivitätsbereich oder weitere physikalische Materialeinheiten, wie T1- und T2-Relaxationszeiten, skaliert werden.
Kontakt
-

Dr. Jörg Peter
Arbeitsgruppenleiter