Computational and Single-Cell Epigenomics
Wer wir sind
Die Forschungsgruppe „Computational and Single-Cell Epigenomics“ ist Teil der Abteilung Krebsepigenomik am Deutschen Krebsforschungszentrum (DKFZ). Wir arbeiten an computergestützten Lösungen für die Untersuchung von Veränderungen im epigenomischen Muster im Zusammenhang mit Krebs, mit einem besonderen Fokus auf das DNA-Methylierungsmusters. Zusätzlich nutzen wir Einzelzell-DNA-Methylierungstechnologien, um Methylierungsveränderungen in seltenen Zellpopulationen zu untersuchen.
Mitarbeitende
- Profil anzeigen

Dr. Michael Scherer
Group Leader Computational and Single-Cell Epigenomics
-

Sergio Manzano Sanchez
Technician
-
Maxime de Vrieze
Projekte
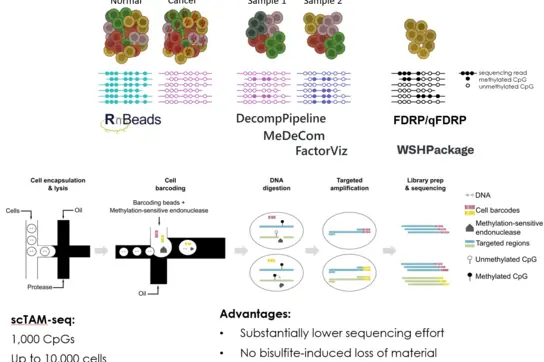
Wir setzen computergestützte Werkzeuge zur Untersuchung epigenetischer Dysregulationen bei Krebs ein bzw. entwickeln sie weiter. Darüber hinaus verwenden wir Einzelzell-Epigenomtechniken (z. B. scTAM-seq), um die epigenetische Heterogenität einzelner Zellen in Krebsproben näher zu untersuchen.
Angebote
Wir sind ständig auf der Suche nach neuen Mitarbeitern, die unser Team verstärken. Zurzeit haben wir die folgenden offenen Stellen:
Bachelor-/Masterarbeitsprojekte im Bereich Computational und Single-Cell Epigenomics
Wir bieten eine Vielzahl von Projekten an, die von der Entwicklung von Computertools über die Datenanalyse bis hin zu Projekten mit Arbeiten im Nasslabor reichen. Derzeit bieten wir die folgenden Projekte an:
- Datenanalyseaufgaben zu verschiedenen Krebsarten unter Verwendung der RnBeads (rnbeads.org/) toolsuite
- Generierung eines zielgerichteten Panels von ca. 1.000 CpGs, die mit scTAM-seq untersucht werden sollen, um epigenetische Heterogenität bei akuter myleoischer Leukämie zu untersuchen
- Datenanalyse von scTAM-seq-Daten und Einzelzell-RNA-seq-Daten
Durch die Einbettung in die Abteilung für Krebsepigenomik bieten wir eine angenehme Arbeitsatmosphäre mit einer Mischung aus Bioinformatikern und experimentellen Studenten.
Was wir suchen: Wir suchen Studenten, die ihren Bachelor- oder Masterabschluss an der Universität Heidelberg machen und ihre Bachelor-/Masterarbeit oder ein Praktikum im Bereich Computational Epigenomics absolvieren möchten. Vorkenntnisse in der Programmierung in R und in der Epigenomik sind von Vorteil.
Publikationen
- Scherer M, Singh I, Braun M, Szu-Tu C, Kardorff M, Rühle J, et al. Somatic epimutations enable single - cell lineage tracing in native hematopoiesis across the murine and human lifespan. bioRxiv. 2024;1–36. https://doi.org/10.1101/2024.04.01.587514
- Bianchi, A., Scherer, M., Zaurin, R., Quililan, K., Velten, L., & Beekman, R. (2022). scTAM-seq enables targeted high-confidence analysis of DNA methylation in single cells. Genome Biology, 23(1), 229. https://doi.org/10.1186/s13059-022-02796-7
- Scherer, M., Nebel, A., Franke, A., Walter, J., Lengauer, T., Bock, C., Müller, F., & List, M. (2020). Quantitative comparison of within-sample heterogeneity scores for DNA methylation data. Nucleic Acids Research, 48(8), e46–e46. https://doi.org/10.1093/nar/gkaa120
- Müller, F., Scherer, M., Assenov, Y., Lutsik, P., Walter, J., Lengauer, T., & Bock, C. (2019). RnBeads 2.0: comprehensive analysis of DNA methylation data. Genome Biology, 20(1), 55. https://doi.org/10.1186/s13059-019-1664-9
- Scherer, M., Nazarov, P. V., Toth, R., Sahay, S., Kaoma, T., Maurer, V., Vedeneev, N., Plass, C., Lengauer, T., Walter, J., & Lutsik, P. (2020). Reference-free deconvolution, visualization and interpretation of complex DNA methylation data using DecompPipeline, MeDeCom and FactorViz. Nature Protocols, 15(10), 3240–3263. https://doi.org/10.1038/s41596-020-0369-6
