Tumorimmunologie und Tumorimmuntherapie
- HI-TRON Mainz
- Immunologie, Infektion und Krebs

Prof. Dr. Niels Halama
Integration breiter Aspekte immunologischer Parameter zur erfolgreichen Entwicklung und Optimierung therapeutischer Ansätze in der klinischen Translation.
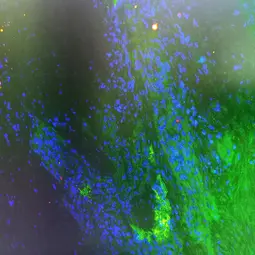
Unsere Forschung
Die Tumorimmunologie hat wissenschaftliche Erkenntnisse erfolgreich in die klinische Praxis überführt und Standards in der Onkologie verändert. Ein tieferes Verständnis der immunologischen Prozesse hat dabei eine erfolgreiche klinische Anwendung ermöglicht. Die Abteilung Tumorimmunologie und Tumorimmuntherapie integriert verschiedene Aspekte immunologischer Parameter (z.B. gewebespezifische Regulation, das (Gewebe-) Mikrobiom, metabolische Regulation, genomische Faktoren u.a.) für die erfolgreiche Entwicklung und Optimierung neuer therapeutischer Ansätze. Ein wichtiger Ansatzpunkt ist ein besseres Verständnis der Regulierung von Immunreaktionen im Gewebe solider Tumoren. Die dedizierte Biomarker-Forschung zur Identifizierung relevanter Immunzellphänotypen ist ebenfalls ein entscheidender Faktor, genau wie das Verständnis der Regulation der Immunantwort durch Zytokin-Netzwerke in bestimmten Krankheitssituationen. Tiefere Einblicke in die Situation von Patienten ermöglicht dabei unser eigens entwickeltes, vollständig humanes, präklinisches Explant-Modellsystem, das die Tumormikroumgebung einzelner Patienten vor Augen führen kann. Darüber hinaus ermöglichen neue, computergestützte Modellsysteme, Datenbanken und maschinelles Lernen („künstliche Intelligenz“) ein besseres Verständnis der Regulierung von Immunantworten. Insbesondere unsere Explant-Modellsysteme eröffnen neue Möglichkeiten bei der Entwicklung neuer Therapien. Neu entwickelte Therapien wurden dabei erfolgreich in die klinische Anwendung überführt. Derzeit wird die kombinierte Immunmodulation basierend auf adaptiven klinischen Studienprotokollen mit biomarkergesteuerten Auswahlprogrammen implementiert.
Die Immuntherapie hat die Onkologie revolutioniert. Eine breite Anwendung immuntherapeutischer Methoden ist bei der Behandlung von Patienten mit hämatologischen und soliden Tumoren bei einigen Krebsarten Standard geworden. Auch im Rahmen des Helmholtz Instituts für Translationale Onkologie (HITRON) wollen wir gemeinsam mit den Partnern Patienten für einen spezifischen immunmodulatorischen Ansatz sowie eine erfolgreiche kombinatorische Immuntherapie (oder Kombination mit Chemotherapie, Bestrahlung oder anderer Intervention wie zellulärer Therapie) identifizieren. Die Verwendung neuer Modellsysteme und informativer Versuchspläne sowie die Verbindung von Grundlagen- und translationaler Forschung machen diesen Bereich sehr vielfältig und herausfordernd, zeigen aber das Potenzial für eine Zukunft der personalisierten Therapie in der Onkologie.
Forschung in Bildern
-

Machine learning Technologien -

Anspruchsvolle Mikroskopie mit der RAMAN-Spektroskopie -

Färberoboter -

Vollständig kontrolliertes Zellkultursystem -

Gewebezüchtung mit dem 3D-Drucker -

Gewebeverarbeitung -
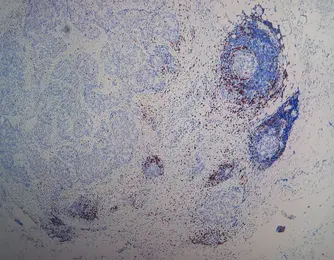
Gescanntes Gewebebild -
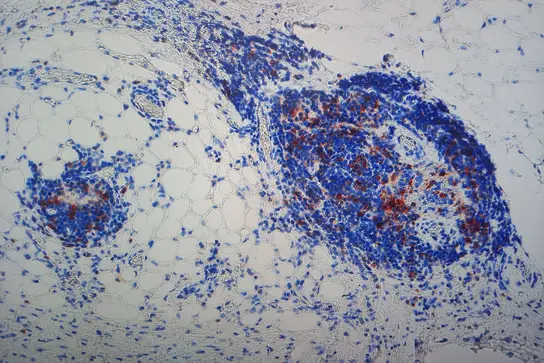
Infiltrierende CD3+-T-Zellen -

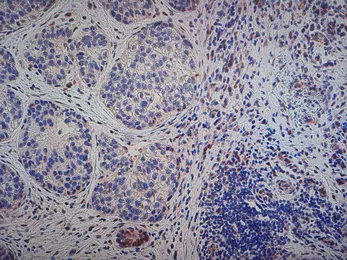
Tumorgewebe mit Fettgewebe -

Kombinatorische Therapiestratifizierung
Projekte
Die langjährige Erkenntnisgewinnung aus der Verknüpfung von Ergebnissen präklinischen Modellsystemen mit frühen klinischen Studien hat zahlreiche mögliche Interventionsmöglichkeiten aufgedeckt und frühe klinische Studien ergeben. Zwei Beispiele für diesen systematischen Ansatz und die erfolgreiche Umsetzung umfassen die Modulation der Chemokinachse CCL5 und des entsprechenden Rezeptors CCR5 (Halama N et al, 2016, Cancer Cell) sowie die Hemmung des Zytokins SDF1alpha. Systematische Analysen der Tumormikroumgebung unter Verwendung des Explant-Modellsystems (Halama N et al, 2016, Cancer Cell) ergaben mögliche Synergien für eine klassische Checkpoint-Immuntherapie in Kombination mit einer Hemmung von CCR5. Die entsprechende Studie (PICASSO, ClinicalTrials.gov-Kennung: NCT03274804) mit begleitenden seriellen Biopsien wurde durchgeführt um die Auswirkungen der Hemmung von Anti-PD-1 (Pembrolizumab) und CCR5 bei massiv vorbehandelten Patienten mit Dickdarmkrebs zu untersuchen. Obwohl die Ergebnisse der PICASSO-Studie veröffentlicht sind, läuft aktuell noch die parallele Studie zur Untersuchung einer dreifachen Immuntherapie-Kombinationsbehandlung (LUMINESCENCE, ClinicalTrials.gov-Kennung: NCT04721301, PI Niels Halama) mit 50 rekrutierten Patienten (25 mit Dickdarmkrebs und 25 mit Bauchspeicheldrüsenkrebs, alle mikrosatellitenstabil). Die Kombination von zwei Checkpoint-Inhibitoren (Anti-PD-1, Anti-CTLA4) mit CCR5-Hemmung weist vielversprechende synergistische Effekte auf, indem die antitumorale Wirkung der Makrophagen-Repolarisierung gesteigert und gleichzeitig eine verstärkte Aktivierung des adaptiven Immunsystems bewirkt wird. Systematische Analysen von Serienbiopsien und des Mikrobioms der Studienpatienten zeigen vielversprechende Ergebnisse, da insbesondere bei etwa 20 % der Patienten die Krankheit unter Kontrolle ist, was diesen Probensatz in einem ansonsten auf Immuntherapie nicht ansprechenden Patiententyp einzigartig macht. Die klinische Verlaufskontrolle und begleitende Forschung laufen noch. Ein weiterer Bestandteil des Tumormikromilieus wird von Fibroblasten gebildet, die eine Barriere für die T-Zell-Infiltration bilden und antitumorale Immunreaktionen beeinträchtigen. Es wurde berichtet, dass die Hemmung von SDF1alpha zur Modulation von Fibroblasten führt und somit die Tumormikroumgebung verändert. Der neue Ansatz der Hemmung spezifischer Zytokine durch ein RNA-Molekül („Spiegelmer-Technologie“) wurde im Rahmen der OPERA-Studie (KEYNOTE-559, ClinicalTrials.gov-Kennung: NCT03168139, PI Niels Halama) angewandt und die Ergebnisse im Journal of Immunotherapy of Cancer veröffentlicht. Hier konnte die Wirkungsweise der SDF1alpha Inhibition aufgeklärt werden und zeigt eine Aktivierung des adaptiven Immunsystems bei einer Untergruppe von Patienten mit einer spezifischen myeloiden Zellpopulation, die vermutlich für die verstärkten Anti-Tumor-Effekte verantwortlich ist.
In diesem Projekt werden die Möglichkeiten algorithmischer Ansätze zur detaillierten Analyse von mikroskopischen Gewebebildern behandelt. Die automatische Klassifizierung von Gewebebildelementen kann auf verschiedene Weise erfolgen wie z.B. systematische Analyse und Optimierung ausgewählter Merkmale, Optimierung des algorithmischen „Backbones“ (ResNet, AlexNet usw.) und Training und Validierung des Convolutional Neural Network (CNN).
Die Analyse von Patientenkohorten mit primären Kolorektalkarzinomen sowie Prognosevorhersagen wurden durchgeführt und in PLOS Medicine veröffentlicht (Kather JN et al, 2018, PLOS Medicine). Diese Veröffentlichung war die am häufigsten zitierte Publikation des Jahres 2019 von PLOS Medicine. Der Ansatz wurde dann weiterentwickelt zur automatischen Identifikation von mikrosatelliteninstabilen, lymphozytenreichen Kolorektalkarzinomen und in Nature Medicine veröffentlicht (Kather JN et al, 2019, Nature Medicine). Aktuelle Forschungsarbeiten mit Algorithmen des maschinellen Lernens zielen darauf ab, eine Plattform zu implementieren, die es ermöglicht, Explant-Modelle und Bioprints (siehe Abschnitt 1.2.4) automatisch zu stratifizieren. Dadurch können die Modellsysteme systematisch gestört und die Änderungen durch maschinelles Lernen automatisch in einer prognostischen Untergruppe eingeordnet werden. Dies kann dann zur Identifizierung neuer möglicher Ziele für Interventionen oder zur Abschätzung der Wirkung bestimmter Medikamente oder Interventionen auf die Tumormikroumgebung eingesetzt werden.
Die Untersuchung spezifischer Krebsuntergruppen und seltener Krebserkrankungen führt zu heterogenen Datensätzen. Solche Datensätze stellen aufgrund ihrer Variabilität und ihres teilweise zusätzlich begrenzten Umfangs erhebliche Herausforderungen für konventionelle maschinelle Lernverfahren dar, da sie die Identifikation konsistenter Muster und das Training robuster Modelle erschweren. Dennoch ist die Interpretierbarkeit der gewonnenen Daten essenziell, um Fortschritte in der Präzisionsmedizin zu sichern. Zur Bewältigung dieser Problematik verfolgen wir zwei Ansätze. Die Konzepte der topologischen Algebra und ihre Überführung in Algorithmen ermöglichen eine präzisere Abbildung der komplexen, tief verwobenen Zusammenhänge biologischer Prozesse (Moraleda et al, 2020, Semin Immunol, Valous et al, 2020 Semin Immunol). Als erster Schritt wurde eine nicht-SQL-basierte Graphdatenbank in Neo4j implementiert, die heterogene und komplexe Daten integriert. Der nächste Schritt umfasst die Entwicklung und Anwendung topologisch basierter Algorithmen, um Datensätze auf neuartige Weise zu clustern und zu klassifizieren. Als ein zweiter Ansatz wurden erste Schritte unternommen, um Quantencomputing-Methoden zu nutzen. Zusammenfassend ermöglicht die Formulierung von Problemen in einem biquadratischen Rahmen die Entwicklung von effizienteren Analyseverfahren. Basierend auf der Quantenannealer-Technologie wird die systematische Analyse von Graphennetzwerken erforscht, und ein erster Prototyp wurde entwickelt, der eine weitere Optimierung mittels geometrischer Algebra ermöglicht, um die Lokalisierung identifizierter zentraler Knoten mit größeren Zweigen des Graphen zu verknüpfen. Diese Untersuchung der Quantenmethodik ist ein gemeinsames Projekt mit Kooperationspartnern vom Fraunhofer IKS (München).
Der Vorteil der Quantenverarbeitung für begrenzte Datensätze wird auch im maschinellen Lernen weiter untersucht, wobei erste Ergebnisse erzielt wurden (Matic A et al., ArXiv, 2022).
Bei der klassischen Immuntherapie steht die Modulation bzw. Aktivierung des adaptiven Immunsystems im Mittelpunkt. Obwohl für manche Tumorerkrankungen exzellente Therapieerfolge erzielt wurden, sprechen andere Tumorerkrankungen nicht auf diese Therapie an. Um das Potenzial der Immuntherapie besser nutzen zu können, forschen wir deshalb an der Rolle der angeborenen Immunität. Da das Mikrobiom einen entscheidenden Einfluss auf den Therapieerfolg haben kann, untersuchen wir die wechselseitige Beziehung zwischen Mikrobiom und angeborener Immunität innerhalb der Tumormikroumgebung. In diesem Rahmen haben wir eine Pipeline auf Basis der Nanopore-Technologie für die 16S-Sequenzierung entwickelt, die eine systematische Definition der Zusammensetzung des Mikrobioms ermöglicht. Weiterhin werden spezifische und hochempfindliche PCR-Assays zur Bestimmung von Schlüsselfaktoren, die das Immunsystem und somit die Patientenprognose beeinflussen, angewendet. Hierzu gehören qPCR-Assays zur Quantifizierung von sowohl Fusobacterium nucleatum als auch dessen Produkten Colibactin, Bacteroides fragilis-Toxin und FadA. Neben der klassischen Mikrobiomanalyse konzentriert sich unsere Forschung auch auf das Mykobiom. Derzeit werden systematische Analysen zur Abgrenzung von Pilzarten auf der Grundlage bestehender Datenbanken wie z. B. UNITE und WARCUP durchgeführt. Erste Ergebnisse der oben beschriebenen Pipeline mit Proben der LUMINESCENCE-Studie (siehe Abschnitt 1.2.1) zeigen vielversprechende entitätsübergreifende Muster, die mit einer guten oder schlechten Prognose bzw. Therapieergebnis korrelieren. Um die Zusammensetzung des Mikrobioms im Gewebe besser zu verstehen, haben wir eine andere Technologie weiterentwickelt: Assembloide oder Bioprints sind künstlich hergestellte Gewebe mit einem definierten Inhalt an Zellpopulationen (z. B. Tumorzellen, T-Zellen, Makrophagen oder Bakterien). Die Matrix bzw. das Gerüst besteht aus einer kollagen- und zytokeratinhaltigen Substanz, in der die gewünschten Zellen eingebettet werden. Die Miniaturisierung ermöglicht einen systematischen Seite-an-Seite Vergleich von unterschiedlichen Bedingungen und Zusammensetzungen. So wird der Effekt von verschiedenen Zytokinmischungen, Bakterien oder Bakteriengemeinschaften auf definierte Populationen von Tumor- und Immunzellen analysiert. Ein erster Anwendungsfall dieses Ansatzes wurde kürzlich veröffentlicht (Ahmed A et al, 2022, Frontiers Immunology).
Team
Ein 14-köpfiges Team bestehend aus Biologen, Molekularbiologen, Physikern und Ärzten arbeitet an der Entwicklung und Optimierung neuer therapeutischer Ansätze zur erfolgreichen Bekämpfung des Krebs.
- Profil anzeigen

Prof. Dr. Niels Halama
-

Dr. Azaz Ahmed
-

Dr. Anna Berthel
-
Dr. Silke Grauling-Halama
-

Florian Heininger
-

Dr. Xu Holtkotte
-

Alicia Höflich
-

Karla Kerekovic
-
Alexandra Pöchmann
-
Dr. Heiko Weyd
-

Silke Will
-
Jana Wolf
-
Xin-Wen Zhang
Ausgewählte Publikationen
Wankhede D, Yuan T, Kloor M, Halama N*, Brenner H, Hoffmeister M
Jahangir CA, Page DB, Broeckx G, … Halama N* et al.
Ahmed A*, Köhler S, Klotz R, Giese N, Hackert, T, Springfeld C, Jäger D, Halama N*
Halama N*, Zoernig I, Berthel A*, Kahlert C, Klupp F, Suarez-Carmona M*, Suetterlin T, Brand K, Krauss J, Lasitschka F, Lerchl T, Luckner-Minden C, Ulrich A, Koch M, Weitz J, Schneider M, Buechler MW, Zitvogel L, Herrmann T, Benner A, Kunz C, Luecke S, Springfeld C, Grabe N, Falk CS, Jaeger D
Kontaktieren Sie uns

Prof. Dr. Niels Halama
AbteilungsleiterPostanschrift:

Marion Drechsel
Office ManagerinPostanschrift:
