Translationale Neuroblastom-Forschung
- Funktionelle und Strukturelle Genomforschung
- KiTZ

Priv. Doz. Dr. Frank Westermann
Abteilungsleiter
Ziel unserer Arbeit ist es, die molekularen Mechanismen bei der Entstehung und Entwicklung von Neuroblastomen zu verstehen und daraus eine präzisere Risikostratifizierung und neue molekular zielgerichtete Therapien zu entwickeln.
Bild: Telomere FISH von CHLA-90 Neuroblastom Zellen,
Bild: Telomere FISH von CHLA-90 Neuroblastom Zellen,
Unsere Forschung
Das Neuroblastom ist eine Krebsart, die ausschließlich bei Kindern auftritt und in den ersten Lebensjahren aus nicht ausgereiften Zellen des peripheren Nervensystems entsteht. Klinisch haben diese Tumoren einen sehr unterschiedlichen Verlauf. Einige Tumoren bilden sich von selbst zurück, während andere sehr aggressiv fortschreiten und zum Tod führen. Neuroblastome, die einen Mechanismus zur Telomerverlängerung aufweisen haben in der Regel eine schlechte Prognose, wohingegen Tumoren mit einer niedrigen Risikoeinstufung meist keinen solchen Mechanismus aufweisen. Unterschiedliche Neuroblastom-Subtypen verlängern Telomere entweder durch Aktivierung des Enzyms Telomerase (als Folge von MYCN-Aktivierung oder TERT-rearrangement) oder durch den alternative lengthening of telomeres (ALT) Mechanismus.
Durch eine umfassende molekulargenetische Charakterisierung der unterschiedlichen Neuroblastom-Subtypen (z. B. mittels globaler Genexpressions-, DNA-Methylierungsanalyse und Hochdurchsatzsequenzierung) möchten wie die ursächlichen genetischen, epigenetischen und metabolischen Veränderungen des Neuroblastoms finden und dessen unterschiedlichen klinischen Verlauf aufklären. Des Weiteren arbeiten wir darauf hin die regulatorischen Netzwerke, die das Epigenom und Transkriptom von Neuroblastom Zellen definieren, besser zu verstehen und ihre Rolle in der Tumorentstehung, Rezidivbildung und Metastasierung zu entschlüsseln. Mit Hilfe von Einzelzellanalysen und räumlich aufgelösten Transkriptionsanalysen (spatial transkriptomics) von Neuroblastom Tumoren und normalen embryonalen und fetalen Geweben verschiedener Entwicklungsstufen des peripheren Nervensystems, untersuchen wir die entwicklungsbiologischen Ursprünge und Entwicklungsverläufe des Neuroblastoms. Unsere (epi)genetischen Arbeiten ergänzen wir durch die Analyse der metabolischen Netzwerke in Neuroblastom-Zellen. Dabei sind wir besonders an der Frage interessiert, ob diese Netzwerke einen Angriffspunkt für neue Therapien darstellen.
Um spezifische Schwachstellen von Neuroblastom-Zellen auszumachen und systematisch Zielstrukturen für die personalisierte Medizin zu identifizieren, nutzen wir funktionelle Hochdurchsatzanalysen (siRNA-/shRNA-Screening-Verfahren und Wirkstoff-Screening-Verfahren). Wir suchen dabei besonders nach Schwachstellen, die mit spezifischen Veränderungen in den Krebszellen assoziiert sind. Wir haben eine langjährige Erfahrung in der präklinischen Entwicklung und Validierung von spezifischen Zielstrukturen und arbeiten dabei eng mit Partnern aus der Klinik und Industrie zusammen.
Projekte
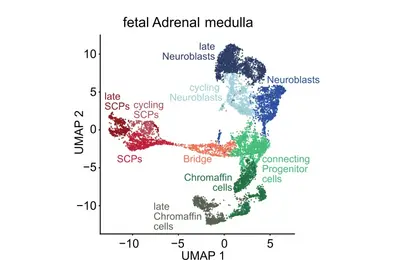
Genomische und andere molekulare Analysen bei verschiedenen Krebsarten haben eine bemerkenswerte Vielfalt an genomischen Aberrationen, veränderten Signalwegen und onkogenen Prozessen aufgezeigt. Wir stellen die Hypothese auf, dass diese Vielfalt durch endogene Faktoren, insbesondere der spezifischen Entwicklungsprogramme und epigenetische Zustände der entstehenden Zellen, in Verbindung mit exogenen Faktoren entsteht. Eine genaue Definition der Ursprungszellen und der Differenzierungs-/Entwicklungszustände während der Schlüsselereignisse in der Tumorentstehung ist von größter Bedeutung. Ziel dieses Forschungsschwerpunkts ist es, Entwicklungsprogramme zu identifizieren, die in Tumorzellen im Vergleich zu normalen embryonalen/fötalen Zellen wieder aktiviert wurden.
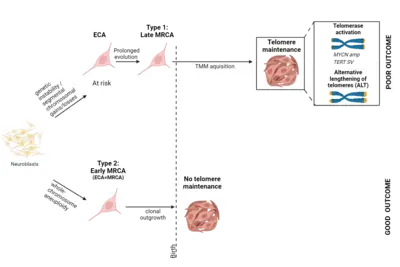
Eine zentrale Frage unserer Arbeit ist, wie bestimmte Mutationen die normale Entwicklung des Ursprungsgewebes verändern. Ziel dieses Forschungsschwerpunktes ist es zu verstehen, wann genau Neuroblastome in der Embryonalentwicklung entstehen und wie sich gutartige von sehr aggressiven Tumoren unterscheiden. Unsere Analysen deuten darauf hin, dass Neuroblastome über das gesamte klinische Spektrum hinweg vermutlich schon während der frühen Schwangerschaft zu entstehen beginnen. Anhand bestimmter Erbgutveränderungen und mit Hilfe von mathematischen Modellen können wir die Entwicklungsgeschichte jedes einzelnen Tumors rekonstruieren. Diese Berechnung basiert auf der Annahme, dass sich Erbgutveränderungen im Genom zufällig und über die Zeit hinweg mit konstanter Geschwindigkeit anhäufen – wie Sand in einer Sanduhr. Die Ansammlung von Mutationen wird daher auch als molekulare Uhr bezeichnet und ist messbar und erlaubt Rückschlüsse auf die zeitliche Entstehungsgeschichte. Die Analysen zeigten überraschenderweise, dass Neuroblastome aller Risikogruppen bereits in der frühen Schwangerschaft entstehen, sich jedoch in Art und Dauer ihrer frühen genetischen Evolution unterscheiden (Körber et al., Nature Genetics 2022).

Um die Entstehung und Entwicklung eines Neuroblastoms besser verstehen zu können, modellieren wir wichtige (epi)genetische Veränderungen, die wir in den Tumoren identifiziert haben, in humanen induzierten pluripotenten Stammzellen (iPSCs). Diese iPSCs können im Labor in verschiedene Entwicklungsstadien der Embryonalentwicklung gezielt differenziert werden. So kann getestet werden in welchen Zellpopulationen bestimmte (epi)genetische Veränderungen zur Entstehung eines Neuroblastoms führen.
Ein wichtiges Ziel unserer Arbeit ist es seit vielen Jahren, Zielmoleküle in Neuroblastomzellen, die von einer hohen Aktivität des Onkogens MYC(N) abhängig sind, zu identifizieren., da MYC(N) selbst nicht oder nur schwer therapeutisch angegriffen werden kann. Auf der Grundlage eines siRNA Screens haben wir die Hemmung des CDK12/13/CCNK Komplex als potenzielle Zielstruktur ermittelt. In einem Wirkstoffscreening haben wir verschiedene chemische Substanzen identifiziert, die spezifisch auf den CDK12/13/CCNK-Komplex abzielen. Diese Substanzen basieren auf einer sehr ähnlichen chemischen Struktur, haben aber unterschiedliche Wirkmechanismen. Einige Substanzen wirken als klassische Kinase Inhibitoren, während andere als sogenannte molekulare „glue degrader“ wirken. Diese Moleküle nutzen das Ubiquitin-Proteasom-System der Zelle um ihre Zielproteine gezielt abzubauen und somit unwirksam zu machen. Unsere Arbeit wird im Rahmen des internationalen Projektes PROTECT als Teil des Programmes „Cancer Grand Challenges“ gefördert. Die entwickelten Substanzen sind klinisch sehr vielversprechend und werden ausführlich präklinisch in Bezug auf Effektivität und Sicherheit getestet.
Ferroptose ist eine Form des Zelltods, der ausgelöst wird, wenn sich eine tödliche Menge an freien Radikalen in der Zelle ansammelt. Dies geschieht, wenn die metabolischen Systeme versagen, die das Gleichgewicht von Sauerstoff, Eisen, Aminosäuren und mehrfach ungesättigten Fettsäuren regulieren. Neuroblastomzellen sind aufgrund ihrer hohen Stoffwechselaktivität besonders anfällig für Ferroptose. Die Krebszellen haben allerdings spezielle Mechanismen entwickelt um sich vor einem Zelltod durch Ferroptose zu schützen. Ziel unserer Arbeit ist es das komplexe regulatorische System in Neuroblastomzellen zu verstehen und dadurch Angriffspunkte zu identifizieren, wie die Ferroptose gezielt ausgelöst werden kann und so neue therapeutische Konzepte entwickelt werden können. Wichtig ist dabei auch zu verstehen, wie sich die regulatorischen Systeme in den verschiedenen molekularen Subtypen des Neuroblastoms unterscheiden und sich daraus eine unterschiedliche Sensitivität für diese Therapien ergibt. Unsere Arbeit zur Ferroptose wird im Rahmen des Schwerpunktprogrammes SPP - 2306 zum Thema "Ferroptose: Von molekularen Grundlagen zu klinischen Anwendungen" von der Deutschen Forschungsgemeinschaft (DFG) gefördert wird.
Unsere Abteilung ist Teil des internationalen Projektes „ITCC pediatric cancer data portal”. Diese internationale Kooperation umfasst klinische Programme aus sieben Nation und hat sich zum Ziel gesetzt genetische Daten von über 6000 pädiatrischen Patienten zu sammeln und zu harmonisieren.
Weiterführende Informationen finden Sie auf der Website des Projektes: https://www.pedcanportal.eu/
Team
Unser Team besteht sowohl aus wet-lab Wissenschaftler:innen als auch Bioinformatiker:innen, sowie technischem Personal.
-

Priv. Doz. Dr. Frank Westermann
Abteilungsleiter
-
Dr. Kai-Oliver Henrich
Senior Wissenschaftler
-
Dr. Sina Kreth
Senior Wissenschaftlerin
-

Dr. Anand Mayakonda Thippeswamy
Leiter Bioinformatik
-
Robin Droit
Postdoc
-

Dr. Sabine Stainczyk
Projektmanagement
-

Cedar Schloo
Doktorand:in
-
Ilayda Özel
Doktorandin
-
Pravin Velmurugan
Doktorand
-

Michael Müller
Doktorand MD/PhD
-
Pascal Kohmann
Medizinischer Doktorand
-
Yeo-Eun Yi
Medizinische Doktorandin
-
Maximilia Eggle
Medizinische Doktorandin
-

Young-Gyu Park
Labor Manager
-

Elisa Maria Wecht
Technische Assistenz - BSc
-
Michelle Müller
Auszubildende Biologielaborantin
-
Adrianna Podolak
Master Studentin
-
Manas Sehgal
Master Student
Ausgewählte Publikationen
Körber, V., S. A. Stainczyk, R. Kurilov, K. O. Henrich, B. Hero, B. Brors, F. Westermann* and T. Höfer*
Alborzinia, H., A. F. Florez, S. Kreth, L. M. Bruckner, U. Yildiz, M. Gartlgruber, D. I. Odoni, G. Poschet, K. Garbowicz, C. Shao, C. Klein, J. Meier, P. Zeisberger, M. Nadler-Holly, M. Ziehm, F. Paul, J. Burhenne, E. Bell, M. Shaikhkarami, R. Wurth, S. A. Stainczyk, E. M. Wecht, J. Kreth, M. Buttner, N. Ishaque, M. Schlesner, B. Nicke, C. Stresemann, M. Llamazares-Prada, J. H. Reiling, M. Fischer, I. Amit, M. Selbach, C. Herrmann, S. Wolfl, K. O. Henrich, T. Höfer*, A. Trumpp*, and F. Westermann*
Single-cell transcriptomic analyses provide insights into the developmental origins of neuroblastoma
Jansky, S., A. K. Sharma, V. Körber, A. Quintero, U. H. Toprak, E. M. Wecht, M. Gartlgruber, A. Greco, E. Chomsky, T. G. P. Grünewald, K.-O. Henrich, A. Tanay, C. Herrmann, T. Höfer, F. Westermann
Hartlieb SA, Sieverling L, Nadler-Holly M, Ziehm M, Toprak UH, Herrmann C, Ishaque N, Okonechnikov K, Gartlgruber M, Park YG, Wecht EM, Savelyeva L, Henrich KO, Rosswog C, Fischer M, Hero B, Jones DTW, Pfaff E, Witt O, Pfister SM, Volckmann R, Koster J, Kiesel K, Rippe K, Taschner-Mandl S, Ambros P, Brors B, Selbach M, Feuerbach L, Westermann F
Gartlgruber M, Sharma AK, Quintero A, Dreidax D, Jansky S, Park Y-G, Kreth S, Meder J, Doncevic D, Saary P, Toprak UH, Ishaque N, Afanasyeva E, Wecht E, Koster J, Versteeg R, Grünewald TGP, Jones DTW, Pfister SM, Henrich K-O, van Nes J, Herrmann C*, Westermann F*
Kontaktieren Sie uns


