Verbundinformationssysteme
- Bildgebung und Radioonkologie
- DKFZ-Hector Krebsinstitut

Prof. Dr. Martin Lablans
Head of Division
Wir bauen Brücken für die biomedizinische Forschung mit neuartigen Methoden, die sowohl technische als auch medizinisch-rechtliche Probleme beim Datenaustausch lösen. Wir wenden diese Methoden an, in realen Netzwerken für die gemeinsame Nutzung von Daten und Bioproben in Europa und darüber hinaus.
Unsere Forschung
Die Erschließung verteilter Daten- und Probenquellen ist daher Voraussetzung für die meisten datengetriebenen Forschungsansätze im Gesundheitswesen. Zudem bildet sie ein vielschichtiges Forschungsfeld zur Entwicklung von Infrastrukturen, die effiziente Forschung in vernetzten Verbünden auf technischer und inhaltlicher Ebene überhaupt erst möglich machen. Auch müssen regulatorische Anforderungen, die sich in der Verarbeitung sensibler Patientendaten naturgemäß ergeben, berücksichtigt werden.
In unserer Abteilung Verbundinformationssysteme untersuchen wir Probleme, die in der vernetzten medizinischen Forschung regelmäßig auftreten:
- Semantik
- Multizentrische Datenintegration aus heterogenen Quellsystemen
- Datenschutz, Einwilligungsmanagement, Record Linkage und Pseudonymisierung
- Verteilte Prozesse zur Auswertung von Daten
- Standortübergreifend vergleichbare Messung der Datenqualität
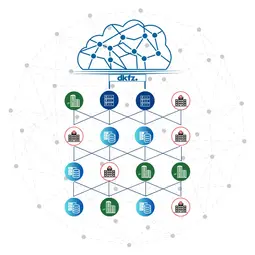
Hierfür entwickeln wir interoperable und nachnutzbare Werkzeuge. Außerdem bringen wir unsere Kenntnisse in nationalen und internationalen Forschungs- und Behandlungsverbünden zum Einsatz. Durch unsere „Brückenköpfe“ erschließen wir ein Netzwerk aus exzellenten Partnern (vgl. Abbildung).
Detaillierte Informationen finden Sie auf unserer englischen Seite
Publikationen
Kontaktieren Sie uns

Prof. Dr. Martin Lablans
Leitung
