Systembiologie der Signaltransduktion
- Funktionelle und Strukturelle Genomforschung

Prof. Dr. Ursula Klingmüller
Abteilungsleiterin
Ein Versagen von Kontrollmechanismen trägt zur Tumorentwicklung bei. Ziel unserer Forschung ist es daher, molekulare Mechanismen zu entschlüsseln, die die Entscheidungen in Zellen kontrollieren und die Kommunikation im Gewebe und zwischen Organen bestimmen.
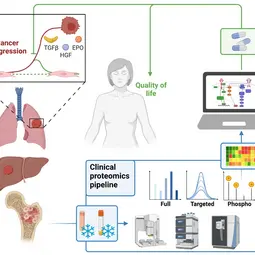
Unsere Forschung

Wir kombinieren quantitative Proteomik mit Mechanismus-basierter mathematischer Modellierung, um eine Früherkennung und personalisierte Behandlung von Krebs zu ermöglichen. Auf diese Weise möchten wir dazu beitragen, die Lebensqualität von Krebspatienten zu verbessern.
Eine Vielzahl von extrazellulären Signalen binden an Rezeptoren, die auf der Zelloberfläche vorhanden sind und die empfangenen Signale in eine Aktivierung von intrazellulären Signalübertragungsnetzwerken umwandeln. Einerseits wird die Dynamik der Signalübertragung in diesen Netzwerken durch den Stoffwechselzustand der Zellen beeinflusst, andererseits hat die Signaltransduktion Auswirkungen auf den Stoffwechsel. Die Reaktionen im Netzwerk werden integriert und in phänotypische Zellreaktionen wie Proliferation, Überleben und Differenzierung umgesetzt. Die meisten der zugrundeliegenden Reaktionen sind nichtlinear und laufen auf sehr unterschiedlichen Zeitskalen ab, die von Minuten bis zu Stunden und Tagen reichen können. Aufgrund dieser Komplexität sind mathematische Modellierungsansätze von Vorteil. Die Entwicklung von Mechanismus-basierten mathematischen Modellen ermöglicht nicht nur die schnelle Prüfung von Hypothesen zu Mechanismen, die zur Krebsentstehung beitragen könnten, sondern erleichtern auch die Vorhersage von Krankheitsverläufen und unterstützen die Entwicklung optimierter Interventionsstrategien zur Therapie bei einzelnen Patienten.
In enger Zusammenarbeit mit Partnern aus der mathematischen Modellierung und klinischen Partnern entwickeln wir Patienten-nahe zelluläre Modellsysteme mit einer authentischen extrazellulären Matrix, entwickeln modernste Massenspektrometrie-Ansätze für die klinische Proteomik und kombinieren zunehmend KI-basierte Modellierungsansätze mit dynamischer Signalwegmodellierung. Im Fokus unserer Arbeiten stehen Prozesse, die die Entwicklung von Leukämie, Lungenkrebs und Leberkrebs bestimmen.
Wesentliche Arbeitsgebiete der Abteilung sind:
- Etablierung von systemmedizinischen Ansätzen für eine klinische Anwendung durch standardisierte Verfahren in der Erzeugung hochwertiger Daten und die Entwicklung prädiktiver mathematischer Modelle
- Mechanistische Aufklärung der durch Erythropoietin (Epo) ausgelösten zellulären Reaktion von der Einzelzelle über die Zellpopulation bis zum gesamten blutbildenden System
- Aufklärung der Interaktionen in der Tumormikroumgebung, die zur Entwicklung von Lungenkrebs beitragen und die für eine optimierte patientenspezifische Therapie verwendet werden können.
- Entschlüsselung der Interaktion von Signaltransduktion und Stoffwechsel, um Mechanismen aufzuklären, die die Kompensation von Leberschäden aufgrund von Medikamenten oder Virusinfektionen steuern, und um modellbasierte Biomarker für die Früherkennung von Leberkrebs zu entwickeln.
Ausblick
Unser Ziel ist es, Mechanismus-basierte mathematische Modelle für die Vorhersage von Krankheitsverläufen zu entwickeln, um eine Früherkennung von Krebserkrankungen zu unterstützen und wirksame Interventionsstrategien zu entwickeln, mit denen ein aggressiv fortschreitender Krebs in eine kontrollierbare Krankheit umgewandelt werden kann. Daher entwickeln wir integrative Modelle, die Signaltransduktion, Stoffwechsel und phänotypische Reaktionen miteinander verknüpfen. Dies sind wesentliche Bausteine für die Erstellung mechanistischer Modelle auf mehreren Ebenen, um die Auswirkungen zellulärer Veränderungen auf das gesamte Organ vorherzusagen. Wir werden unsere auf Massenspektrometrie basierende klinische Proteomik-Pipeline weiterhin standardisieren und automatisieren, um die Erhebung von zuverlässigen quantitativen Daten zu gewährleisten und durch die Kombination von KI-basierter Mustererkennung mit dynamischer Signalwegmodellierung die Entwicklung von Vorhersagemodellen zu ermöglichen. Durch unsere klinischen Partner haben wir Zugang zu hochwertigen Patientenproben, die es uns ermöglichen, mit unseren prädiktiven mathematischen Modellen Krankheitsmechanismen zu entschlüsseln, die einen Übergang von chronischen Erkrankungen zu Krebs begünstigen oder Auswirkungen von Krebstherapien auf das Herz bestimmen.
Projekte
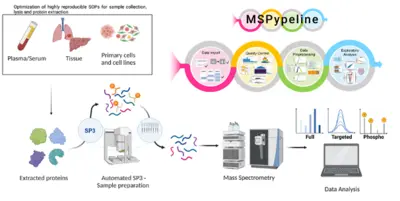
Weiterentwicklung der klinischen Proteomik für die Systemmedizin
Die Entstehung und der Verlauf von Krankheiten sind hochdynamische Prozesse. Um Veränderungen zu identifizieren, die zur Deregulierung führen, sind systemmedizinische Ansätze erforderlich, die hochwertige Datengenerierung mit der Entwicklung mechanistischer mathematischer Modelle kombinieren. Da biologische Funktionen durch Proteine ausgeführt werden und deren Konzentration entscheidend für die zellspezifische Informationsverarbeitung ist, wird ein quantitativer proteomischer Ansatz benötigt, der sowohl für die mathematische Modellierung als auch für die klinische Anwendung robust genug ist.
Eine wichtige Grundlage für eine belastbare, klinisch anwendbare Pipeline ist die standardisierte Sammlung von Patientenproben gemäß Standardarbeitsanweisungen (SOPs) (Wessels et al., Transl Lung Cancer Research 2020; Gegner et al., Front Mol Biosci 2022). Wichtige Schritte unserer klinischen Proteomik-Pipeline umfassen die Optimierung der Zell-, Extrazellulärmatrix-, Gewebe- und Blutplasmalyse, die teilautomatisierte Probenverarbeitung und die Integration von Qualitätsstandards. Zur Standardisierung der Datenverarbeitung und Sicherstellung der Reproduzierbarkeit wurde das MSPypeline-Tool (Heming et al., Bioinform Adv 2022) entwickelt. Mit diesen Fortschritten konnten wir bereits zeigen, dass die generierten Daten die Kalibrierung unserer mechanistisch basierten dynamischen Pfadmodelle für die Signaltransduktion von TGFβ, HGF, EGF, IL-6, TNFα, IFNα und Epo erheblich verbessern.
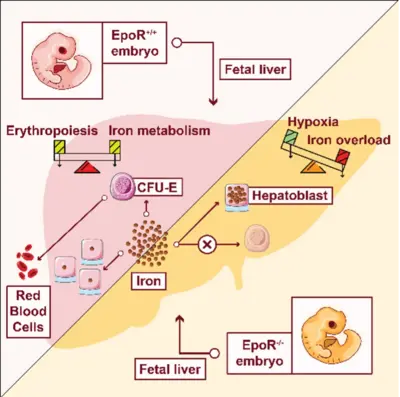
Brücke von der Einzelzelle zur Zellpopulation – Lernen aus Mechanismen, die Erythropoetin (Epo)-induzierte Zellantworten und Erythroleukämie steuern
Ein gesunder Mensch produziert pro Sekunde 2,5 Millionen rote Blutkörperchen (Erythrozyten). Trotz dieser hohen Proliferationsrate ist Erythroleukämie relativ selten. Der Schlüsselregulator der Erythropoese ist das Hormon Erythropoetin (Epo), das von der Niere ausgeschüttet wird und primär auf erythroide Vorläuferzellen im Stadium der Colony-Forming Unit-Erythroid (CFU-E) wirkt. CFU-E-Zellen exprimieren den Epo-Rezeptor (EpoR) und benötigen Epo für ihr Überleben, ihre Proliferation und Differenzierung. Da diese Zellen leicht zugänglich sind und in vitro kultiviert werden können, eignet sich dieses System hervorragend zur Etablierung von Strategien für die Erzeugung hochwertiger Daten und zur Weiterentwicklung mathematischer Modellierungsansätze zur Aufklärung regulatorischer Schlüsselmechanismen. Während auf Populationsebene eine abgestufte Zunahme des Überlebens von CFU-E-Zellen als Reaktion auf Epo-Stimulation beobachtet wird, ist das Überleben auf Einzelzellebene eine binäre Entscheidung. Durch mathematische Modellierung konnten wir zeigen, dass die Heterogenität der Epo-induzierten STAT5-Phosphorylierung auf Variabilität in membranassoziierten Prozessen und im Zytoplasmavolumen zurückzuführen ist und eine Überlebensschwelle definiert. Das zytoplasmatische Volumen der CFU-E-Zellen wandelt also ein schalterartiges Verhalten in einzelnen Zellen in abgestufte Reaktionen auf Populationsebene um (Adlung et al., Cell Rep 2021). In Abwesenheit von EpoR versagt die Hämoglobinproduktion in CFU-E-Zellen, und in der fetalen Leber von EpoR-Knockout-Embryonen wird eine massive Eisenüberladung beobachtet, die mit einer Verringerung der an der oxidativen Phosphorylierung beteiligten Eisen-Schwefel-Cluster-Proteine einhergeht. Daher erweist sich die Verbindung zwischen Erythropoese, Regulierung der Eisenhomöostase und metabolischer Reprogrammierung als entscheidend für den Schutz des Embryos vor Eisenintoxikation und sein Überleben (Chakraborty et al., Cell Rep 2022). Derzeit werden die umfassenden dynamischen Signalwegmodelle, die für die Epo-induzierte Signaltransduktion in CFU-E-Zellen und zellulären Modellsystemen etabliert wurden (Schilling et al., Mol Syst Biol 2009; Becker et al., Science 2010, Bachmann et al., Mol Syst Biol 2011, Adlung et al., Mol Syst Biol 2017), an die Situation in von Patienten stammenden Erythroleukämiezellen angepasst, um Mechanismen zu entschlüsseln, die zum Fortschreiten der Krebserkrankung beitragen, und um vielversprechende Interventionsstrategien vorzuschlagen.
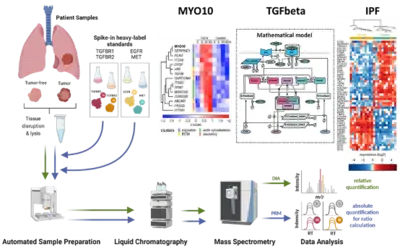
Entschlüsselung von Wechselwirkungen in der Tumormikroumgebung, die die Entwicklung von Lungenkrebs bestimmen
Lungenkrebs ist nach wie vor eine Krankheit mit hoher Inzidenz und hoher Sterblichkeit. Die häufigste Form des nicht-kleinzelligen Lungenkarzinoms (NSCLC) ist das Adenokarzinom der Lunge (LUAD), gefolgt vom Plattenepithelkarzinom (LUSC). Der transformierende Wachstumsfaktor beta (TGFβ) spielt bei Krebs eine doppelte Rolle, da er als Tumorsuppressor entdeckt wurde, aber auch den epithelialen-to-mesenchymalen Übergang (EMT) bei metastasierenden Erkrankungen begünstigt. Da hohe TGFβ-Konzentrationen mit einem schlechten Ergebnis bei Lungenkrebs korreliert sind und TGFβ als Schlüsselfaktor für den Umbau der extrazellulären Matrix (ECM) identifiziert wurde, ist es von besonderer Bedeutung, seine komplexe Rolle in der Tumormikroumgebung bei Lungenkrebs zu entschlüsseln. Wir haben durch phänotypische Studien und zeitaufgelöstes Next Generation Sequencing gezeigt, dass TGFβ die Hochregulierung des Nicht-Muskel-Myosins MYO10 induziert. Interessanterweise zeigten unsere Studien, dass das Expressionsverhältnis von MYO10 im Tumor und im tumorfreien Gewebe prognostisch für das Gesamtüberleben von LUSC-Patienten ist und die Vorhersage des Ansprechens auf eine Chemotherapie erleichtert (Dvornikov et al., Sci Rep 2018). Das Fortschreiten des Tumors hängt jedoch nicht nur von Veränderungen in der Krebszelle ab, sondern wird auch stark von der Kommunikation innerhalb der Mikroumgebung des Tumors beeinflusst. Die Bedeutung der Fibroblasten bei der Entstehung von Lungenkrebs wird durch die Beobachtung unterstrichen, dass Patienten mit idiopathischer Lungenfibrose (IPF) ein erhöhtes Risiko haben, Lungenkrebs zu entwickeln. In alveolären Typ-2-Zellen eines IPF-Mausmodells, das einen konditionalen Knockout der Ubiquitin-Ligase Nedd4-2 aufweist, beobachteten wir eine verstärkte TGFβ-induzierte SMAD-Signaltransduktion und eine erhöhte Expression von Komponenten, die mit dem Umbau der extrazellulären Matrix verbunden sind (Duerr et al., Nature Commun 2020). Derzeit entwickeln wir ein dynamisches Modell der TGFβ-Signaltransduktion, um die Auswirkungen des ECM-Umbaus und der Zell-Zell-Kommunikation auf Lungenkrebszellen quantitativ zu erfassen. Wir untersuchen von Patienten stammende Proben, um mechanismusbasierte Biomarker für die Früherkennung von Lungenkrebs und geschlechtsspezifische Unterschiede aufzudecken.
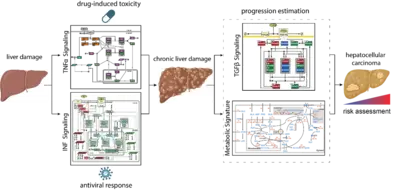
Mechanismen zur Kompensation von Leberschäden und modellbasierte Biomarker zur Früherkennung von Leberkrebs
Die Leber spielt eine zentrale Rolle im Stoffwechsel und der Entgiftung. Da sie ständig schädlichen Einflüssen ausgesetzt ist, verfügt sie über eine bemerkenswerte Regenerationsfähigkeit. Diese Prozesse müssen jedoch streng reguliert werden, um die physiologische Funktion zu erhalten. Wir identifizierten mithilfe modellbasierter Selektion die drei relevantesten SMAD-Komplexe in Leberzellen. Unser mathematisches Modell legt nahe, dass erhöhte SMAD-Proteinspiegel und SMAD2-Phosphorylierung Kennzeichen von Leberkrebs sind (Lucarelli et al., Cell Syst 2018). Der Einfluss von Tumornekrosefaktor Alpha (TNFα) auf die Regulation der Leberregeneration wurde ebenfalls untersucht. Durch dynamische Pfadmodellierung konnten wir zeigen, dass das nichtsteroidale entzündungshemmende Medikament Diclofenac mehrere Parameter im TNFα-Netzwerk beeinflusst und zu einer verstärkten Toxizität führt (Oppelt et al., NPJ Syst Biol Appl 2018).
Neben Medikamenten können auch hepatotrope Viren wie das Hepatitis-B-Virus (HBV) oder das Hepatitis-C-Virus (HCV) Leberschäden verursachen und antivirale Interferon-alpha (IFNα)-Reaktionen auslösen. Durch die dynamische Modellierung der Signalwege konnten wir die molekularen Mechanismen aufklären, die eine Hypersensibilisierung oder Desensibilisierung der Signalwege vermitteln. Die Anpassung des mathematischen Modells für einzelne Patienten in einer virtuellen Patientenkohorte ergab patientenspezifische Prädiktoren für die Desensibilisierung des Signalwegs (Kok et al, Mol Syst Biol 2020). Aufgrund der Schlüsselrolle der Leber im Stoffwechsel ist es von großem Interesse, die Schnittstellen zwischen der Signaltransduktion bei der Leberregeneration und dem Stoffwechsel aufzuklären. In einem ersten Schritt trugen wir zu einem genomweiten Modell bei, das eine hohe Aminosäureverbrauchsrate in Hepatomzellen identifizierte und auf die Bedeutung der Glutamatausscheidung hinwies, die eine mögliche krebsspezifische Intervention darstellt (Nilsson et al., Proc Natl Acad Sci U S A 2020). Gegenwärtig liegt der Schwerpunkt auf der Bedeutung des Umbaus der ECM und des Stoffwechsels für das Fortschreiten des Krebses, und diese Ereignisse werden im Blutplasma der Patienten widergespiegelt, um die Früherkennung von Leberkrebs zu erleichtern, wenn eine chirurgische Resektion mit Heilungsabsicht noch möglich ist.
Anwendung der dynamischen Modellierung für die personalisierte Medizin
Die Leber spielt eine zentrale Rolle im Stoffwechsel und bei der Entgiftung. Da das Organ ständig schädigenden Einflüssen ausgesetzt ist, verfügt es über eine bemerkenswerte Fähigkeit zur Regeneration und zur Sicherung des Überlebens. Um die physiologische Funktion aufrechtzuerhalten, erfordern diese Reaktionen strenge Regulierungsmechanismen. Im Gegensatz zu Ansätzen des maschinellen Lernens, die große Datensätze erfordern, können mechanistische Modelle auf der Grundlage kleiner Datensätze kalibriert werden und als prädiktive Werkzeuge eingesetzt werden, um eine kausale Beziehung zwischen Inputs und Outputs herzustellen (D'Alessandro et al, Biochem J 2022). TGFβ ist ein wichtiger Kontrollfaktor, der die Beendigung der Leberregeneration koordiniert. Durch modellbasierte Auswahl haben wir die drei wichtigsten SMAD-Komplexe in Leberzellen identifiziert. Dieses mathematische Modell legt nahe, dass erhöhte Spiegel von SMAD-Proteinen und die SMAD2-Phosphorylierung Kennzeichen von Leberkrebs sind (Lucarelli et al., Cell Syst 2018). Ein weiterer Faktor, der in die Regulierung der Leberregeneration involviert ist, ist der Tumor-Nekrose-Faktor alpha (TNFα). Wir haben durch dynamische Pfadmodellierung gezeigt, dass mehrere Parameter im TNFα-Netzwerk durch den nichtsteroidalen Entzündungshemmer Diclofenac beeinflusst werden, was zu einer verstärkten Toxizität führt und die Toxizitätsschwelle definiert (Oppelt et al. NPJ Syst Biol Appl 2018). Neben Medikamenten können auch hepatotrope Viren wie das Hepatitis-B-Virus (HBV) oder das Hepatitis-C-Virus (HCV) Leberschäden verursachen und antivirale Interferon-alpha (IFNα)-Reaktionen auslösen. Durch die dynamische Modellierung der Signalwege konnten wir die molekularen Mechanismen aufklären, die eine Hypersensibilisierung oder Desensibilisierung der Signalwege vermitteln. Die Anpassung des mathematischen Modells für einzelne Patienten in einer virtuellen Patientenkohorte ergab patientenspezifische Prädiktoren für die Desensibilisierung des Signalwegs (Kok et al, Mol Syst Biol 2020). Aufgrund der Schlüsselrolle der Leber im Stoffwechsel ist es von großem Interesse, die Schnittstellen zwischen der Signaltransduktion bei der Leberregeneration und dem Stoffwechsel aufzuklären. In einem ersten Schritt trugen wir zu einem genomweiten Modell bei, das eine hohe Aminosäureverbrauchsrate in Hepatomzellen identifizierte und auf die Bedeutung der Glutamatausscheidung hinwies, die eine mögliche krebsspezifische Intervention darstellt (Nilsson et al., Proc Natl Acad Sci U S A 2020). Gegenwärtig liegt der Schwerpunkt auf der Bedeutung des Umbaus der ECM und des Stoffwechsels für das Fortschreiten des Krebses, und diese Ereignisse werden im Blutplasma der Patienten widergespiegelt, um die Früherkennung von Leberkrebs zu erleichtern, wenn eine chirurgische Resektion mit Heilungsabsicht noch möglich ist.
Team
- Profil anzeigen

Prof. Dr. Ursula Klingmüller
Abteilungsleiterin
-
Yomn Abdullah
Doktorandin
-
Annalisa Addante
Postdoktorandin
-
Sandra Bonefas
-
Dr. Sebastian Burbano De Lara Carrillo
Postdoktorand
-
Katharina Büchner
-
Simone Clas
Doktorandin
-

Kyra Fischer
-
Franziska Gödtel
-
Barbara Helm
-
Elisa Holstein
Doktorandin
-
Martina Kegel
-
Daniel Kempter
Masterstudent
-
Nicola Kunz
-

Dr. Nantia Leonidou
Postdoktorandin
-
Caroline Lohoff
-
Till Möcklinghoff
-
Christina Mölders
Doktorandin
-
Benedikt Niedermaier
-
Akane Ogawa
Gast-Bachelorstudentin
-

Dr. Agustin Rodriguez Gonzalez
Postdoktorand
-
Tobias Roth
-
Michelle Sadlowski
-

Dr. Marcel Schilling
Stellvertreter
-
Lisa Strotmann
-
Katerina Sulková
Doktorandin
-
Cong Quan Ta
Doktorand
-
Florian Tichawa
-
Leonie Wilhelm
-

Dr. Piotr Zadora
Postdoktorand
-
Waheb Hasan Zaidi
Ausgewählte Publikationen
Sebastian Burbano de Lara, Svenja Kemmer, Ina Biermayer, Svenja Feiler, Artyom Vlasov, Lorenza A D’Alessandro, Barbara Helm, Christina Mölders, Yannik Dieter, Ahmed Ghallab, Jan G Hengstler, Christiane Körner, Madlen Matz-Soja, Christina Götz, Georg Damm, Katrin Hoffmann, Daniel Seehofer, Thomas Berg, Marcel Schilling, Jens Timmer, and Ursula Klingmüller
Sajib Chakraborty, Geoffroy Andrieux, Philipp Kastl , Lorenz Adlung, Sandro Altamura, Martin E. Boehm, Luisa E. Schwarzmüller, Yomn Abdullah, Marie-Christine Wagner, Barbara Helm, Hermann-Josef Gröne, Wolf D. Lehmann, Melanie Boerries, Hauke Busch, Martina U. Muckenthaler, Marcel Schilling, and Ursula Klingmüller.
Lorenz Adlung, Paul Stapor, Christian Tönsing, Leonard Schmiester, Luisa E Schwarzmüller, Lena Postawa, Dantong Wang, Jens Timmer, Ursula Klingmüller, Jan Hasenauer, and Marcel Schilling.
Disentangling molecular mechanisms regulating sensitization of interferon alpha signal transduction.
Frédérique Kok, Marcus Rosenblatt, Melissa Teusel, Tamar Nizharadze, Vladimir Gonçalves Magalhães, Christopher Dächert, Tim Maiwald, Artyom Vlasov, Marvin Wäsch, Silvana Tyufekchieva, Katrin Hoffmann, Georg Damm, Daniel Seehofer, Tobias Boettler, Marco Binder, Jens Timmer, Marcel Schilling, and Ursula Klingmüller.
Alle Publikationen
Kontaktieren Sie uns





