RNA-Protein Komplexe und Zellproliferation
- Funktionelle und Strukturelle Genomforschung

Priv. Doz. Dr. Maiwen Caudron-Herger
Einmal pro Zellzyklus stehen Zellen vor der anspruchsvollen Aufgabe, ihre Chromosomen auf zwei identische Sätze zu verteilen. Obwohl die Zellteilung stark reguliert wird, können dabei Fehler auftreten, die entweder zum Zelltod oder zur Genominstabilität führen. Der Genominstabilität liegen menschlichen Krankheiten wie z.B. Krebserkrankungen zugrunde. RNA-Protein-Komplexe haben sich als kritische regulatorische Elemente in wichtigen zellulären Prozessen erwiesen, auch während der Zellteilung.

“Zellen haben die wunderbare Fähigkeit, eine Vielzahl dynamischer Strukturen zusammenzubauen, die sehr komplexe und spezifische Aufgaben erfüllen. Unser Ziel ist es, unser Verständnis der zugrunde liegenden Regulierungsmechanismen zu verbessern und die Frage, wie Funktionsstörungen zu Krankheiten führen können, zu beantworten.”
PD Dr. Maïwen Caudron-Herger
Unsere Forschung
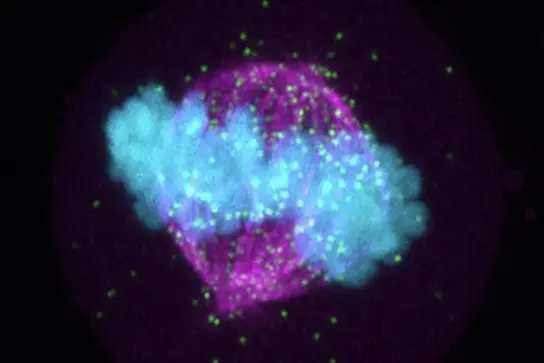
Trotz erheblicher Fortschritte in der Zellbiologie sind die komplexen Prozesse, die die Zellteilung regulieren und vorantreiben, immer noch nicht vollständig verstanden. Dies bietet Möglichkeiten für Entdeckungen im Zusammenhang mit den Ursachen und Anfälligkeiten stark proliferativer Krankheiten wie Krebs.
RNA-Protein-Komplexe haben sich hierbei als entscheidende regulatorische Elemente in mehreren wichtigen zellulären Prozessen mit erheblicher Bedeutung für die Gesundheit und Krankheit herausgestellt. Dies macht sie zu attraktiven Zielen für die Forschung.
Unsere aktuellen Projekte befassen sich mit der zentralen Rolle von RNA und RNA-Protein-Komplexen bei der Koordination der funktionellen Zusammenstellung kritischer mitotischer Strukturen für die fehlerfreie Vollendung der Zellteilung. Durch die Integration der RNA-Komponente verleiht unser Ansatz der Analyse wichtiger Ereignisse in dieser grundlegenden Phase des Zellzyklus ein neues Maß an Komplexität.
Unser Ansatz zeichnet sich durch eine interdisziplinäre und kollaborative Herangehensweise aus. Er umfasst ein vielfältiges Spektrum an Methoden. Dazu gehören Immunpräzipitation zur Untersuchung von Protein-Protein-Wechselwirkungen, auf UV-Vernetzung und Immunpräzipitation (iCLIP) basierende Ansätze zur Untersuchung von Protein-RNA-Wechselwirkungen, Hochdurchsatzsequenzierung und bioinformatische Analysen zur Identifizierung interagierender RNA-Transkripte, sowie konfokale Mikroskopie-Bildgebung zur Lokalisierung von Proteinen, RNAs und ihrer Interaktionen.
Neben den Auswirkungen auf unser grundlegendes Verständnis der Zellteilung hat das neu erworbene Wissen großes Potenzial in innovative Krebstherapie-Strategien umgesetzt zu werden, indem es noch unerforschte zelluläre Funktionen in den Fokus nimmt, die mit spezifischen RNA-Protein-Wechselwirkungen verbunden sind.
Unser Team
-

Priv. Doz. Dr. Maiwen Caudron-Herger
Group Leader
-

Dr. Simona Cantarella
Postdoc
-

Jeanette Simone Seiler
Technical Assitant
-

Jana Theiß
Technical Assistant
-

Jinxin Wang
Erasmus Student
-

Jule Neffe
Student - Master Biochemistry
-

Jamie Kleer
Student - Molecular Biotechnology - Bachelor Thesis
Unsere Projekte
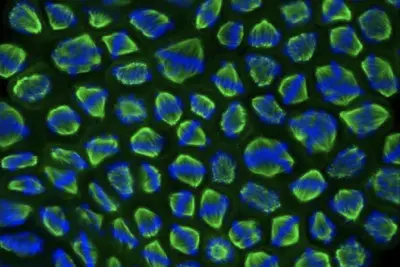
Wir haben einen Atlas RNA-abhängiger Proteine erstellt (siehe Konzept unten), der eine große Anzahl mitotischer Faktoren als unkonventionelle RNA-bindende Proteine hervorhebt: Sie scheinen in Abwesenheit bekannter RNA-bindender Domänen an RNA zu binden. Dies deutet auf eine wichtige Rolle der RNA während der Zellteilung in mehreren Teilen der Zellteilungsmaschinerie (Mikrotubuli, kondensierte Chromosomen, Kinetochoren, Zentrosomen usw.) hin, die wir besser verstehen wollen.
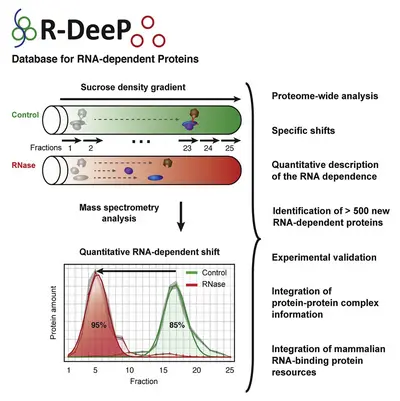
Wir haben kürzlich das Konzept der RNA-Abhängigkeit entwickelt. Dabei definieren wir ein Protein als RNA-abhängig, wenn es nur unter Anwesenheit von RNA mit anderen Proteinen interagiert oder ein Komplex bildet. Dieses Konzept wurde in eine Methode namens R-DeeP umgesetzt, welche umfassend und unvoreingenommen proteomweite und quantitative Screenings durchführt.
R-DeeP basiert auf der Fraktionierung von Zelllysat – mit und ohne vorherige RNase-Behandlung – durch Saccharose-Dichtegradienten-Ultrazentrifugation und anschließender Analyse durch proteomweite Massenspektrometrie oder Western Blot einzelner Proteine.
R-DeeP bestimmt die Fähigkeit eines Proteins, Proteinkomplexe ausschließlich in Gegenwart von RNA zu bilden, entweder durch direkte oder indirekte Interaktion mit den RNA-Molekülen. Im R-DeeP-Screening haben wir eine signifikante Anreicherung von Mikrotubuli-bezogenen Proteinen festgestellt. Dies weist auf bislang unbekannte mögliche funktionelle Auswirkungen von RNA-Protein-Interaktionen hin, insbesondere im Zusammenhang mit der Zellteilung.
Besuchen Sie gerne unsere aktualisierte Datenbank unter R-DeeP3.dkfz.de.
Die Analyse von RNA-Protein-Komplexen ist von zentraler Bedeutung für die Aufklärung der komplexen molekularen Netzwerke, die zelluläre Prozesse steuern. Mit der zunehmenden Anerkennung ihrer Bedeutung haben Proteine, die direkt mit RNA interagieren, wachsendes Interesse geweckt. Die umfassende, aber spezifische Identifizierung solcher RNA-bindenden Proteine (RBPs) und die Analyse ihrer Funktionen in Verbindung mit RNA bleiben jedoch große Herausforderungen auf dem Gebiet der RNA-Biologie.
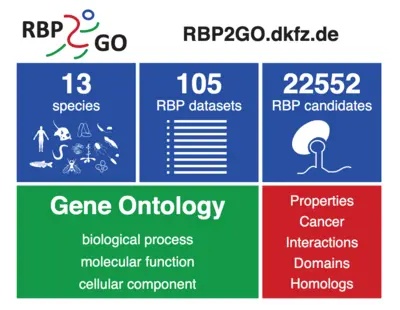
Um unser Wissen über RBPs weiter zu vertiefen, wurden mehrere proteomweite Strategien entwickelt, um RBPs in verschiedenen Spezies zu identifizieren. Demnach hat eine große Anzahl von Studien eine Reihe von Katalogen bereitgestellt, die experimentell identifizierte und vorhergesagte RBP-Kandidaten auflisten. Die rasante Entwicklung des Fachgebiets hat zu einer Anhäufung isolierter Datensätze geführt, was sowohl die Zugänglichkeit als auch die Vergleichbarkeit erschwert hat. Darüber hinaus haben Werkzeuge zur Verknüpfung von RBPs mit zellulären Signalwegen und Funktionen gefehlt. Angetrieben durch unsere eigene Forschung und als Service für die wissenschaftliche Gemeinschaft haben wir die Datenbank RBP2GO entwickelt, eine umfassende Datenbank aller verfügbaren proteomweiten Datensätze für RBPs für 13 verschiedene Arten, die einen Katalog von 22552 RBP-Kandidaten bereitstellt (RBP2GO.DKFZ.de). Es umfasst nicht nur Daten zu den Interaktionspartnern jedes RBPs, sondern liefert auch Informationen zu den biologischen Prozessen, molekularen Funktionen und zellulären Komponenten in denen dieses involviert ist.
RBP2GO bietet eine benutzerfreundliche Weboberfläche. Der RBP2GO Score, ein neu eingeführtes Bewertungssystem, ermöglicht es dem User die Eignung potenzieller RBP-Kandidatenen als echte RBPs einzuschätzen. Die “Erweiterte Suche” von RBP2GO bietet Benutzern zudem Optionen zum Navigieren der miteinander verbundenen Datensätze, um neue Forschungsrichtungen zu fördern und anzuregen.
Laufende Projekte verfeinern derzeit die Liste der RBPs durch die Einbeziehung von Informationen zu RNA-Bindungsdomänen (RBDs) und RNA-verwandten Proteinfamilien. Diese Mehrzweckanalysen zielen darauf ab, die umfangreiche Liste der RBP-Kandidaten zu verfeinern und auf der Grundlage eines Bewertungssystems eine Liste hochzuverlässiger RBPs bereitzustellen (RBP2GO-2-beta.dkfz.de).
Unsere Datenbanken

RBP2GO bietet eine umfassende Datenbank von RNA-bindenden oder RNA-abhängigen Proteinen aus allen verfügbaren proteomweiten Studien in 13 verschiedenen Arten. Sie enthält die Annotation ihrer Funktionen sowie Interaktionspartner - und ermöglicht auch die umgekehrte Suche nach RNA-bindenden Proteinen mit spezifischen molekularen Funktionen, biologischen Prozessen, zellulären Kompartimenten oder einer bekannten Verbindung zu Krebs. RBP2GO wurde in Nucleic Acids Reseach 2021 veröffentlicht. Eine neue Version von RBP2GO ist jetzt online verfügbar (RBP2GO-2-beta), die Informationen über die Domänen der Proteine und insbesondere über die RNA-bindenden Domänen bietet, wie in Nucleic Acids Research 2024 veröffentlicht.

R-DeeP3 liefert die proteomweiten Screening-Ergebnisse für RNA-abhängige Proteinkomplexe aus unsynchronisierten HeLa S3- und A549-Lungenkrebszellen sowie aus synchronisierten HeLa-Zellen in Mitose und Interphase. Darüber hinaus bietet R-DeeP3 Optionen für den Vergleich aller Datensätze und für den Download der Ergebnisse.
R-DeeP liefert die proteomweiten Screening-Ergebnisse für RNA-abhängige Proteinkomplexe auf der Grundlage unseres Artikels in Molecular Cell 2019. Es enthält den vollständigen Datensatz aus HeLa S3-Zellen und bietet zusätzliche Suchoptionen für Proteinkomplexe und RBPs aus verschiedenen Quellen.
Ausgewählte Publikationen
Rajagopal V, Seiler J, Nasa I, Cantarella S, Theiß J, Herget F, Kaifer B, Klostermann M, Will R, Schneider M, Helm D, König J, Zarnack K, Diederichs S, Kettenback AN and Caudron-Herger M.
Wassmer E, Koppány G, Hermes M, Diederichs S and Caudron-Herger M.
Caudron-Herger M*, Ralf E. Jansen, Elsa Wassmer and Sven Diederichs. *Corresponding author.
Caudron-Herger M*, Barreau E, Nasa I, Schultz A, Seiler J, Kettenbach AN and Diederichs S*. *Corresponding authors.
Caudron-Herger M*, Rusin SF, Adamo ME, Seiler J, Schmid V, Barreau E, Kettenbach AN and Diederichs S*. *Corresponding authors.
Lehre an der Universität Heidelberg
Fakultät für Ingenieurwissenschaften
- Methoden der Bioinformatik: Datenanalyse und Bildanalyse (BSc Molekulare Biotechnologie, Wintersemester)
- Anwendung bioinformatischer Systeme: Datenanalyse (BSc Molekulare Biotechnologie, Sommersemester)
Fakultät für Biowissenschaften
- Grenzgebiete der Biowissenschaften: Molekulare, genetische und bioinformatische Ansätze in der Krebsforschung (Major Krebsbiologie, Wintersemester)
- Physikalische Methoden in der Systembiologie (Studiengang Systembiologie, Sommersemester)
Seminar
- Gestaltung einer wissenschaftlichen Präsentation - Visuelle Kommunikation für Wissenschaftler

Kontaktieren Sie uns
