Multiparametrische Methoden zur Früherkennung des Prostatakarzinoms
- Bildgebung und Radioonkologie
- Klinische Kooperationseinheit
- Nachwuchsgruppe

Priv. Doz. Dr. Magdalena Görtz
Gruppenleitung
Unsere im Jahr 2021 etablierte Nachwuchs-Klinische Kooperationseinheit am DKFZ und der Urologischen Universitätsklinik Heidelberg widmet sich klinischen Fragestellungen und treibt die patientenorientierte translationale Forschung im Bereich des Prostatakarzinoms voran. Unsere primären Ziele sind die individualisierte Früherkennung und Risikostratifizierung mit besonderem Fokus auf die Identifizierung aggressiver Prostatakarzinome zur Optimierung von Diagnostik und Therapieplanung.
Bild: © ©Magdalena Görtz/dkfz.de

Bild: © ©Magdalena Görtz/dkfz.de
Unsere Forschung
Prostatakrebs stellt aufgrund seiner unterschiedlichen Aggressivität und Progression eine große Herausforderung dar. Dies unterstreicht den dringenden Bedarf an neuen molekularen Markern und integrierten Diagnosemodellen, die eine zuverlässige Unterscheidung zwischen aggressiven und indolenten Prostatakarzinomen sowie gutartigen Erkrankungen ermöglichen.
Wir haben prospektiv eine umfassende Datenkohorte und Biobank aufgebaut, die Arztbriefe, Laborbefunde, MRT-Bildgebung, digitale Pathologie, (epi)genomische, transkriptomische und proteomische Daten sowie das onkologische Follow-Up der Patienten umfasst. Durch die Integration verschiedener Datenmodalitäten (klinische Parameter, Labor- und Bildgebungsdaten sowie molekulare Flüssigkeits- und Gewebedaten) und durch den Einsatz hochmoderner Computertechnologien zielen wir auf eine integrative Einschätzung des Krankheitsstatus jedes einzelnen Patienten, um die Früherkennung und Risikostratifizierung zu verbessern. Die Extraktion relevanter Features aus multimodalen Daten und deren Integration in ein auf maschinellem Lernen basierendes Vorhersagemodell ermöglicht eine umfassende Darstellung des Tumors des Patienten bei Erstdiagnose und damit eine Personalisierung von Diagnose- und Therapieentscheidungen. Eine integrative Strategie zur Früherkennung und Risikostratifizierung, die klinische Parameter, molekulare Marker und bildgebende Verfahren kombiniert, hat das Potenzial, die nicht-invasive Diagnostik zu optimieren, präzisere Vorhersagemodelle zu entwickeln und Korrelationen mit der Aggressivität des Prostatakarzinoms zu ermöglichen.
Im Rahmen unseres starken translationalen Ansatzes, der auf klinische Auswirkungen abzielt, arbeiten wir in Konsortialprojekten mit akademischen und industriellen Partnern wie der Universität Heidelberg und Siemens Healthineers zusammen, um KI-basierte Lösungen zur Entscheidungsunterstützung und zur „similar-patient search“ zu entwickeln. Diese Partnerschaften stellen eine Brücke zwischen klinischer Fragestellung, akademischer Forschung und Industrie dar und stellen sicher, dass innovative diagnostische und therapeutische Ansätze einen direkten Einfluss auf die Patientenversorgung haben. Entsprechend unserer patientenzentrierten Ausrichtung haben wir in Zusammenarbeit mit SAP SE einen KI-basierten Chatbot entwickelt, um Patienten über die state-of-the-art Früherkennung von Prostatakrebs zu informieren. Dieses validierte KI-Tool ist ein Beispiel für unser Bestreben, aktuelle Entwicklungen des maschinellen Lernens in relevante klinische Anwendungen zu bringen.
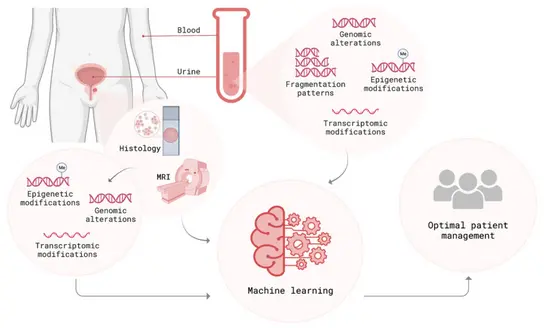
Forschungsprojekte
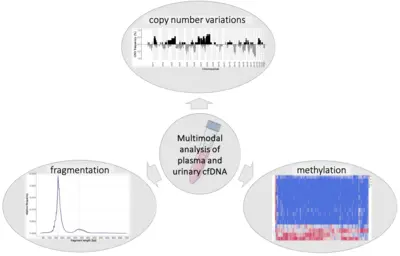
Wir untersuchen prädiktive (epi)genetische Veränderungen in zirkulierender zellfreier DNA von Prostatakrebspatienten als nicht-invasive Methode zur Unterscheidung von aggressiven Tumoren von indolentem Prostatakrebs und gesunden Patienten.
Die multimodale Analyse von molekularen Markern in Blut und Urin für die nicht-invasive Diagnostik birgt ein großes Potenzial für die Weiterentwicklung der Früherkennung und Risikostratifizierung von Prostatakrebs. Die zirkulierende, vom Tumor stammende DNA spiegelt den heterogenen molekularen Status des Primärtumors und seiner Metastasen wider. Unsere Forschungsgruppe hat einen Workflow zur Analyse von Methylierungsmustern, Fragmentierungsprofilen und Kopienzahlveränderungen in zellfreier DNA etabliert, der auf Flüssigbiopsieproben von neu diagnostizierten Prostatakrebspatienten und tumorfreien Kontrollen basiert. Die multimodale Analyse birgt vielversprechendes Potential bei der Krebsfrüherkennung, da sie Synergien zwischen verschiedenen Datentypen nutzt. Unsere Ziele sind eine präzise molekulare Charakterisierung und eine adäquate Bewertung der Tumoraggressivität bei der Erstdiagnose von Prostatakrebs, um optimale, risikoangepasste Therapieentscheidungen zu ermöglichen.
Kooperation:
Abteilung „Krebsgenomforschung“, DKFZ
HI-STEM Nachwuchsgruppe „Pattern Recognition and Digital Medicine“
Early Cancer Institute, Abteilung für Onkologie, Universität Cambridge

Wir integrieren klinische Daten, Routineblutparameter und Metabolomics, um nicht-invasive Strategien für die Früherkennung von aggressivem Prostatakrebs zu entwickeln.
Wir evaluieren prospektiv, ob die Integration klinischer Daten mit Routineblutparametern die nicht-invasive Früherkennung von aggressivem Prostatakrebs verbessern kann. Unsere Forschung hat neue Blutmarker identifiziert, die in Kombination mit konventionellen PSA-Tests und klinischen Parametern die diagnostische Genauigkeit signifikant erhöhen können. Ein daraus entwickeltes mehrstufiges Risikomodell übertrifft reine PSA-Methoden und bietet einen kostengünstigen, nicht-invasiven und personalisierten Ansatz für die Früherkennung. Mit Hilfe von breit verfügbaren und kostengünstigen Parametern identifiziert unser Modell Patienten mit hohem Prostatakrebsrisiko, die weitere diagnostische Maßnahmen benötigen.
Parallel dazu analysieren wir das Metabolom von Patienten als additive Strategie zur Früherkennung von aggressivem Prostatakrebs. Durch den Vergleich von metabolischen Profilen in Blut- und Urinproben wollen wir relevante Unterschiede in den Metabolitenkonzentrationen zwischen Patienten mit aggressivem Prostatakrebs, indolenter Erkrankung und gesunden Personen identifizieren.
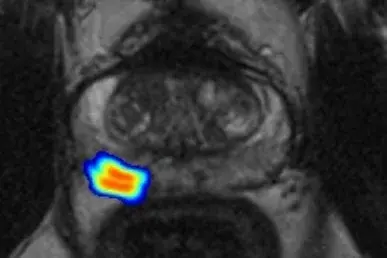
Durch die Extraktion quantitativer Features aus der MRT-Bildgebung, die Integration mit klinischen und molekularen Daten und die Anwendung maschinellen Lernens werden Korrelationen zwischen bildgebenden Befunden und der zugrunde liegenden Tumorbiologie detektiert.
Angesichts der begrenzten Sensitivität und Spezifität der derzeitigen Methoden zur Früherkennung von Prostatakrebs ist die Integration mehrerer Datenmodalitäten entscheidend für eine präzise Diagnose. Unsere Gruppe konzentriert sich daher auf die Kombination von multimodalen Daten, um die Entscheidungsfindung bei der Diagnostik und Therapie von Prostatakrebs zu optimieren. Dies umfasst die Integration klinischer und bildgebender Daten für individualisierte Früherkennungs- und Follow-Up-Strategien sowie die Optimierung der MRT-Ultraschall-Fusion bei der Prostatabiopsie.
Um die nicht-invasive Früherkennung von Prostatakrebs zu verbessern und unnötige Prostatabiopsien zu reduzieren, wenden wir Deep Learning auf bildgebende Daten an und integrieren diese mit klinischen und molekularen Daten. Durch die Kombination von klinischen und bildgebenden Befunden mit molekularen Daten aus Flüssigbiopsien zielen wir auf präzisere, nicht-invasive Diagnosestrategien ab. Aus MRT-Bildgebung extrahierte radiologische Features dienen als Input für maschinelle Lernalgorithmen und werden mit Fragmentierungsprofilen und Markern genomischer Instabilität aus Flüssigbiopsieproben korreliert. Diese Integration von klinischen Daten, radiologischen Merkmalen und molekularen Markern kann nicht nur die nicht-invasive Risikoeinschätzung von Prostatakrebs verbessern, sondern auch Einblicke in Genotyp-Phänotyp-Korrelationen ermöglichen.
Kooperation:
Abteilung Radiologie, DKFZ
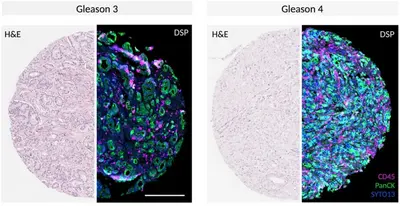
Mit Hilfe der räumlichen Transkriptomik und Proteomik charakterisieren wir die Tumormikroumgebung mit hoher Auflösung und identifizieren Marker für aggressiven Prostatakrebs bei der Erstdiagnose.
Dieses Projekt untersucht Prostatakrebs im Frühstadium mithilfe räumlicher Transkriptomik- und Proteomik-Verfahren, darunter modernste Einzelzell-Imaging-Plattformen. Dadurch gewinnen wir einen hochaufgelösten räumlichen Einblick in die Heterogenität der Prostatatumorzellen und ihrer intratumoralen Mikroumgebung. Um diese räumlichen Datensätze zu analysieren, werden Algorithmen des maschinellen Lernens und der Multi-Omics-Datenintegration verwendet. Diese Methoden helfen uns, wichtige molekulare Interaktionen und Signalwege zu identifizieren, die die Aggressivität des Tumors steuern. Unser Ziel ist es, diese Erkenntnisse in klinisch relevante Stratifizierungsmodelle zu integrieren, die eine präzise Risikoabschätzung und personalisierte Behandlungsempfehlungen für Patienten mit Prostatakrebs im Frühstadium ermöglichen. Die prospektive Studienkohorte der Arbeitsgruppe, die umfassende klinische Informationen, onkologisches Follow-Up und Bildgebung umfasst, bietet eine ideale Umgebung für die Umsetzung sowohl molekularer als auch klinischer Erkenntnisse.
Kooperation:
Abteilung „Bioinformatik der Genomik und Systemgenetik“, DKFZ
Pathologisches Institut, Universitätsklinikum Heidelberg
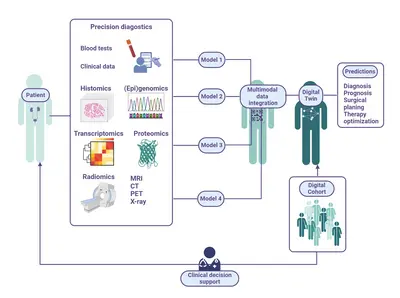
Unser Ziel ist es, molekulare, bildgebende und klinische Daten zu einem „digitalen Zwilling“ von Prostatakrebspatienten zu integrieren und dabei KI einzusetzen, um patientenspezifische, multidimensionale Gesundheitsdaten zu erfassen und reale Tumoreigenschaften widerzuspiegeln.
Seit vielen Jahren arbeiten wir mit Partnern aus Akademie und Industrie zusammen, mit klarem Fokus auf Präzisionstherapie und klinischer Translation. Diese Zusammenarbeit ermöglichte es uns, kompetitiv Drittmittel für die translationale Prostatakrebsforschung beim Bundesministerium für Wirtschaft und Klimaschutz einzuwerben. Die Nachwuchs-Klinische Kooperationseinheit war ein integraler Bestandteil des CLINIC 5.1 Konsortiums (Konsortialleitung). Dieses Konsortium brachte das Deutsche Krebsforschungszentrum, das Universitätsklinikum Heidelberg, die Universität Heidelberg, KARL STORZ, SAP SE und die Siemens Healthineers AG zusammen, um innovative, marktorientierte KI-basierte Entscheidungshilfen für Ärzte zu entwickeln. Der Fokus des Konsortiums liegt auf der KI-basierten Verbesserung der Entscheidungsfindung und Präzisionsmedizin bei der Diagnose, Behandlungsplanung von Prostatakrebs. Das Ziel des Projektes CLINIC 5.1 ist es, durch die Integration und Erweiterung diagnostischer und therapeutischer Datensätze eine umfassende, vierdimensionale virtuelle Darstellung von Prostatakrebspatienten („digitale Zwillinge“) zu entwickeln.
Darüber hinaus wurde eine Zusammenarbeit mit dem Engineering Mathematics and Computing Lab (Interdisciplinary Center for Scientific Computing, Universität Heidelberg) begonnen, um die multimodale Analyse von Prostatakrebs voranzutreiben. Das Projekt hat drei Hauptziele: die Verwendung von partiellen Differentialgleichungen zur Modellierung des Tumorwachstums, die Entwicklung eines multimodalen KI-Frameworks zur Schätzung der Wahrscheinlichkeit eines Tumorrezidivs und die Sicherstellung der Interpretierbarkeit multimodaler KI-Modelle, um das Vertrauen in die KI-Modelle zu stärken. Ein wesentliches Ziel dieser Zusammenarbeit ist das Vorantreiben von Präzisionsonkologie durch innovative, multidisziplinäre Ansätze mit einer soliden mathematischen Grundlage.
Team
Ein engagiertes, interdisziplinäres Team von Experten aus den Bereichen Medizin, Molekularbiologie, Biotechnologie und Bioinformatik arbeitet gemeinsam daran, die nicht-invasive Früherkennung von Prostatakrebs voranzutreiben und die Risikostratifizierung bei der Erstdiagnose zu optimieren, um individualisierte Therapieentscheidungen für die Patienten zu ermöglichen.
- Profil anzeigen

Priv. Doz. Dr. Magdalena Görtz
Gruppenleitung
-

Dr. Anja Lisa Riediger
PhD Studentin
-

Dr. Martina Heller
Clinician Scientist
-

Isabella Schindler
Research Assistant (M.Sc.)
-

Daniela Janscho
Study Nurse
Optimierung der Früherkennung von Prostatakrebs: Die Vision der Arbeitsgruppe
In diesem Video erläutert PD Dr. Magdalena Görtz die Bedeutung eines individualisierten Ansatzes bei der Früherkennung von Prostatakrebs und stellt innovative Methoden vor, die ihr Forschungsteam entwickelt, um die Behandlung von Patienten zu optimieren.

Förderer
Wir sind dankbar für die finanzielle Unterstützung der folgenden Förderinstitutionen, die uns die Durchführung unserer Forschungsprojekte ermöglichen:

Ausgewählte Publikationen
Eickelschulte S, Kaczorowski A, Janke F, Riediger AL, Lazareva O, Böning S, Kristiansen G, Schwab C, Stenzinger A, Sültmann H, Duensing S, Duensing A, Görtz M
Lazareva O, Riediger A, Stegle O, Sültmann H, Hohenfellner M, Görtz M
Baumgärtner K, Byczkowski M, Schmid T, Muschko M, Woessner P, Gerlach A, Bonekamp D, Schlemmer HP, Hohenfellner M, Görtz M
Janke F, Stritzke F, Dvornikovich K, Franke H, Angeles AK, Riediger AL, Ogrodnik S, Gerhardt S, Regnery S, Schröter P, Bauer L, Weusthof K, Görtz M, Harrabi S, Herfarth K, Neelsen C, Paech D, Schlemmer HP, Abdollahi A, Adeberg S, Debus J, Sültmann H, Held T.
Schrader A, Netzer N, Hielscher T, Görtz M, Zhang KS, Schütz V, Stenzinger A, Hohenfellner M, Schlemmer HP, Bonekamp D
Lleshi E, Milne-Clark T, Lee Yu H, Martin HW, Hanson R, Lach R, Rossi SH, Riediger AL, Görtz M, Sültmann H, Flewitt A, Lynch AG, Gnanapragasam VJ, Massie CE
Görtz M, Baumgärtner K, Schmid T, Muschko M, Woessner P, Gerlach A, Byczkowski M, Sültmann H, Duensing S, Hohenfellner M
Eickelschulte S, Riediger AL, Angeles AK, Janke F, Duensing S, Sültmann H, Görtz M
Tavakoli AA, Hielscher T, Badura P, Görtz M, Kuder TA, Gnirs R, Schwab C, Hohenfellner M, Schlemmer HP, Bonekamp D
Görtz M, Hohenfellner M
Görtz M, Radtke JP, Hatiboglu G, Schutz V, Tosev G, Guttlein M, Leichsenring J, Stenzinger A, Bonekamp D, Schlemmer HP
Weitere Publikationen
Kontaktieren Sie uns
