Krebsgenomforschung
- Funktionelle und Strukturelle Genomforschung

Prof. Dr. Holger Sültmann
Leitung
Die Evolution von Krebs ist mit weitreichenden molekularen Veränderungen des Genoms, des Epigenoms und der Expression von Genen verbunden. Die Ziele der Abteilung sind die Identifizierung dieser molekularen Marker, sie für die Diagnose, Prognose und Vorhersage des Therapie-Ansprechens bei Krebspatienten einzusetzen und ihre Rolle bei der Entstehung und Progression von Tumoren zu verstehen.
Bild: Forschungsinhalte Abteilung Krebsgenomforschung am DKFZ, © DKFZ, Abteilung Krebsgenomforschung
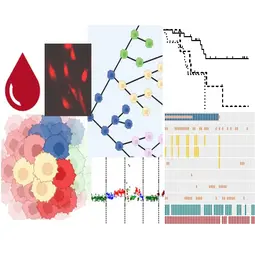
Bild: Forschungsinhalte Abteilung Krebsgenomforschung am DKFZ, © DKFZ, Abteilung Krebsgenomforschung
Unsere Forschung
Unsere Forschung umfasst hauptsächlich die Genom-weite Analyse molekularer Veränderungen in verschiedenen Tumoren mit den Schwerpunkten Lungen- und Brustkrebs. Hierbei nutzen wir Hochdurchsatz-Technologien zur DNA- und RNA-Sequenzierung in Gewebe und Blutproben („Liquid Biopsy“) für die individuelle Risikoabschätzung und das klinische Management der Patient*Innen auf der Basis von molekularen Markern der Tumorheterogenität in Raum und Zeit. Um die Rollen dieser Faktoren bei der Tumorprogression und Therapieresistenz zu verstehen, verwenden wir zelluläre 3D-Kokultur-Modelle sowie eine Vielzahl von zell- und molekularbiologischen Methoden.
Krebszellen unterscheiden sich von normalen Zellen durch ihre Mikroumgebung, veränderte (epi)genomische Merkmale sowie Gen- und Proteinexpression. Die Charakterisierung solcher Veränderungen in Tumoren und verschiedenen Tumorstadien ist entscheidend, um die Mechanismen der Tumorprogression zu verstehen und molekulare Marker für Diagnose und Prognose zu definieren. Die räumliche und zeitliche Heterogenität von Tumoren bestimmt den Therapieerfolg und das Überleben der Patienten. Daher konzentrieren wir uns auf präzisionsmedizinische Ansätze in der Krebsforschung um folgende zentrale Fragen zu adressieren:
Risikostratifizierung von Tumoren mithilfe multimodaler Biomarker
Mechanismen der Tumorprogression und des Therapieversagens
Vorhersage des Therapieansprechens durch nichtinvasive Ansätze
Projekte
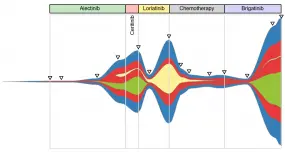
Liquid Biopsy zur nicht-invasiven Tumorüberwachung
Das molekulare Tumor-Profiling ist für die klinische Praxis essentiell, da die genomische Zusammensetzung eines Tumors erheblichen Einfluss auf sein Ansprechen auf eine Therapie hat. Typischerweise wird eine histopathologische Analyse von Gewebeproben durchgeführt, um die molekularen Eigenschaften eines Tumors zu bestimmen. Gewebe-Biopsien haben jedoch auch Nachteile: Sie sind invasiv, spiegeln möglicherweise nicht die gesamte Tumorheterogenität wider und liefern kaum Informationen über Metastasen.
Im Gegensatz zu einer herkömmlichen Biopsie nutzt die Flüssigbiopsie (Liquid Biopsy) Nukleinsäuren, Proteine oder andere Moleküle, die aus Tumoren in Körperflüssigkeiten wie Blut oder Urin abgegeben werden. Die Entnahme solcher Flüssigkeiten ist bereits Teil der medizinischen Routine, minimal invasiv und mit nur geringem Risiko für den Patienten verbunden. Ein weiterer Vorteil der Liquid Biopsy ist, dass das Tumormaterial nicht nur vom Primärtumor stammt, sondern auch von Metastasen, was wertvolle Einblicke in die Tumorheterogenität ermöglicht.
Wir konzentrieren uns auf die Anwendung der Liquid Biopsy, um:
- neue genomische Biomarker zur zeitlichen Kontrolle von metastasiertem Brustkrebs zu identifizieren (SATURN3)
- epigenomische (DNA-Methylierung) und genomische Veränderungen (einzelne Nukleotid-Varianten und Kopienzahl-Varianten) zu analysieren, um minimale Resterkrankungen und Therapieresistenz zu erkennen
- Änderungen der klonalen Zusammensetzung von Tumoren mithilfe molekularer Veränderungen in longitudinalen Blutproben nachzuverfolgen
- das Ansprechen auf eine Therapie durch Analyse von Fragmentierungsmustern der zellfreier DNA vorherzusagen
Weitere Quellen:
Tumoren mittels Blutprobe auf der Spur: https://www.youtube.com/watch?v=rpGLAdQBCdk
Methods for ctDNA detection and analysis: https://youtu.be/pwK1BTlQl6w
Kooperationen:
- Abt. Thoraxonkologie, Thoraxklinik Heidelberg
- Abt. Gynäkologische Onkologie, NCT Heidelberg
- Abt. Molekulare Genetik, DKFZ Heidelberg
- KKE Multiparametrische Methoden zur Früherkennung des Prostatakarzinoms, DKFZ und Universitätsklinikum Heidelberg
- Abt. Radioonkologie, Universitätsklinikum Heidelberg
- Pathologisches Institut, Universitätsklinikum Heidelberg
- Klinik für Hämatologie und Onkologie, Universitätsklinikum Schleswig-Holstein, Lübeck
Unterstützung: Bundesministerium für Bildung und Forschung (BMBF) in den Förderprogrammen Deutsches Zentrum für Lungenforschung (DZL), Deutsches Konsortium für Translationale Krebsforschung (DKTK), OUTLIVE-CRC, und SATURN3
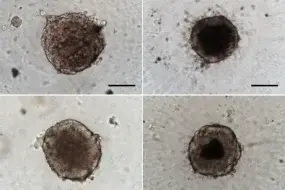
Modellierung der räumlichen Heterogenität und Therapieresistenz bei Lungenkrebs
Tumore zeigen eine erhebliche Heterogenität in ihrer zellulären Zusammensetzung, weshalb es entscheidend ist, die Tumorumgebung, einschließlich der krebsassoziierten Fibroblasten (CAFs), im Kontext des Tumorverlaufs und der Therapieantwort zu berücksichtigen. Um den Einfluss von CAFs auf die Reaktion der Tumorzellen auf bestimmte Medikamente zu untersuchen, haben wir 3D-Co-Kulturmodelle von nicht-kleinzelligem Lungenkrebs („non-small cell lung cancer“, NSCLC) mit CAFs entwickelt. Durch eine Kombination von Einzelzell-RNA-Sequenzierung (scRNA-seq), epigenomischem Profiling, massenspektrometrischer Untersuchung von Proteom und Metabolom stellten wir fest, dass von CAF-sekretierte Moleküle den Lipidstoffwechsel in Tumorzellen mit ALK-Translokationen beeinflussen. Darüber hinaus identifizierten wir neuartige Ligand-Rezeptor-Interaktionen zwischen CAFs und Tumorzellen.
Unsere derzeitige Forschung konzentriert sich auf die Untersuchung, wie die Tumorumgebung, einschließlich CAFs und Immunzellen, Stoffwechselwege im NSCLC verändert und die Therapieresistenz beeinflusst.
Kooperationen: Thoraxklinik Heidelberg; Pathologisches Institut des Universitätsklinikums Heidelberg
Unterstützung: Bundesministerium für Bildung und Forschung (BMBF) im Förderprogramm Deutsches Zentrum für Lungenforschung (DZL).
Team
Die Mitarbeitenden in der Abteilung besitzen Erfahrungen in Molekular- und Zellbiologie, Medizin oder KI-gestützten Datenanalysen. Unser Ziel ist die Erforschung von Therapieresistenz und die Translation von molekularen Markern in die klinische Diagnostik.
-

Prof. Dr. Holger Sültmann
Leitung
-

Dr. Isabell Berneburg
-

Priv. Doz. Dr. Petros Christopoulos
-

Simay Dolaner
-

Sabrina Gerhardt
-

Dr. Kate Glennon
-
Myesha Jahin
-

Dr. Florian Janke
-

Priv. Doz. Dr. Sabine Klauck
-
Dr. Astrid Laut
-

Simon John Ogrodnik
-

Dr. Rebecca Schunk
-
Dr. Andrey Turchinovich
Ausgewählte Publikationen
Janke, F, Gasser, M, Angeles, AK, Riediger, AL, Görtz, M, Appenheimer, L, Laut, AK, Ogrodnik, S, Gerhardt, S, Stenzinger, A, Schneider, MA, Thomas, M, Christopoulos, P, Sültmann, H
Angeles AK, Janke F, Daum AK, Reck M, Schneider MA, Thomas N, Christopoulos P, Sültmann H.
Janke F, Angeles AK, Riediger AL, Bauer S, Reck M, Stenzinger A, Schneider MA, Muley T, Thomas M, Christopoulos P, Sültmann H.
The ICGC/TCGA Pan-Cancer Analysis of Whole Genomes Consortium.
Kontaktieren Sie uns







