Bei der Auswertung medizinischer Bilddaten verspricht die künstliche Intelligenz (KI) Unterstützung und Entlastung für die Ärzte – insbesondere auch in der Krebsmedizin. Doch ob es darum geht, die Ausdehnung eines Hirntumors für die Therapieplanung zu vermessen oder die Rückbildung von Lungenmetastasen im Verlaufe einer Strahlentherapie zu dokumentieren: Zunächst müssen die Rechner die dreidimensionalen Bilddatensätze der CTs oder MRTs zu interpretieren lernen. Sie müssen unterscheiden können, welcher Bildpunkt zum Tumor gehört und welcher nicht. Diese Unterscheidung bezeichnen KI-Experten als semantische Segmentierung.
Für jede einzelne Aufgabenstellung – etwa ein Nierenkarzinom in CT-Bildern oder einen Brustkrebs im MRT zu erkennen – müssen Wissenschaftler spezielle Algorithmen entwickeln, die diese Unterscheidung vornehmen und Vorhersagen treffen können. Als Trainingsmaterial für das maschinelle Lernen dienen dabei Bilddatensätze, in denen Ärzte bereits per Hand markiert haben, was Tumor ist, was gesundes Gewebe und was andere wichtige anatomische Strukturen sind.
Die Entwicklung solcher Segmentierungs-Algorithmen erfordert Erfahrung und Expertenwissen. „Das ist nicht trivial und geht normalerweise mit zeitaufwändigem ‚Versuch und Irrtum' einher“, sagt Medizininformatiker Fabian Isensee, einer der Erstautoren der aktuellen Publikation. Zusammen mit seinen Kollegen in der Abteilung von Klaus Maier-Hein am DKFZ hat er nun eine Methode entwickelt, die sich dynamisch und vollautomatisch beliebigen Bilddatensätzen anpasst und so auch Personen mit geringeren Vorkenntnissen ermöglicht, selbstlernende Algorithmen für bestimmte Aufgabenstellungen zu konfigurieren.
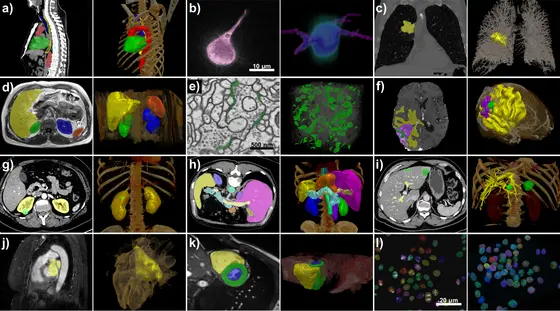
Das nnU-Net genannte Verfahren kann mit einer großen Bandbreite an Bilddaten umgehen: Neben den klassischen bildgebenden Verfahren wie CT und MRT lassen sich damit auch Bilder aus der Elektronen- und Fluoreszenzmikroskopie bearbeiten.
In internationalen Fachwettbewerben erzielten die DKFZ-Forscher mit nnU-Net bei 33 von 53 verschiedenen Segmentierungsaufgaben den besten Platz – obwohl sie dabei gegen hochspezifische und von Experten für individuelle Spezialfragen entwickelte Algorithmen antraten.
Das Team um Klaus Maier-Hein stellt nnU-Net als Open-Source-Tool kostenfrei zum Download zur Verfügung. „nnU-Net ist sofort einsatzbereit, kann auf Bilddatensätzen trainiert werden und spezielle Aufgaben erfüllen – ohne dass dafür besondere computerwissenschaftliche Spezialkenntnisse oder eine außergewöhnliche Rechner-Ausstattung erforderlich wäre“, erklärt Klaus Maier-Hein.
Bislang wird die KI-unterstützte Auswertung medizinscher Bilddaten hauptsächlich im Forschungskontext genutzt und hat noch keinen breiten Einzug in die klinische Routineversorgung von Krebspatienten gehalten. Doch Medizininformatiker und Ärzte sehen großes Anwendungspotenzial, beispielsweise wenn es um hochrepetitive Aufgaben geht, wie sie häufig im Rahmen großer klinischer Studien auftreten. „nnU-Net kann dabei helfen, dieses Potenzial zu heben“, so Studienleiter Maier-Hein.
Fabian Isensee, Paul F. Jaeger, Simon A. A. Kohl, Jens Petersen, and Klaus H. Maier-Hein: nnU-Net: A Self-Configuring Method for Deep Learning-based Biomedical Image Segmentation.
Nature Methods 2020, DOI: 10.1038/s41592-020-01008-z
Ein Bild zur Pressemitteilung steht zum Download zur Verfügung:
figure1_final-.jpg
BU: nnU-Net verarbeitet eine Vielzahl von Datensätzen und Bildeigenschaften. Alle Beispiele stammen aus den Testsätzen verschiedener internationaler Segmentierungs-Wettbewerbe. Die Zielstrukturen für jeden Datensatz wurden in 2D auf die Rohdaten projiziert (links) und außerdem zusammen mit einem Volumen-Rendering der Rohdaten in 3D dargestellt (rechts).
Nutzungshinweis für Bildmaterial zu Pressemitteilungen
Die Nutzung ist kostenlos. Das Deutsche Krebsforschungszentrum (DKFZ) gestattet die einmalige Verwendung in Zusammenhang mit der Berichterstattung über das Thema der Pressemitteilung bzw. über das DKFZ allgemein. Bitte geben Sie als Bildnachweis an: „Quelle: Isensee et al. / Nature Methods“.
Eine Weitergabe des Bildmaterials an Dritte ist nur nach vorheriger Rücksprache mit der DKFZ-Pressestelle (Tel. 06221 42 2854, E-Mail: presse@dkfz.de) gestattet. Eine Nutzung zu kommerziellen Zwecken ist untersagt.