Theoretische Systembiologie
- Funktionelle und Strukturelle Genomforschung

Prof. Dr. Thomas Höfer
Wir untersuchen, wie Krebs in sich entwickelnden oder erneuernden Geweben entsteht. Zu diesem Zweck entwickeln wir prädiktive computergestützte Modelle der somatischen Evolution, der Stammzelldynamik und der Immunantworten.
Bild: Computergestützte Rekonstruktion der Evolution von Neuroblastomen durch Genomsequenzierung (Schema adaptiert aus G. Caravagna, Mathematical modeling of neuroblastoma associates evolutionary patterns with outcomes, Nat. Genet. 2023)., © dkfz.de
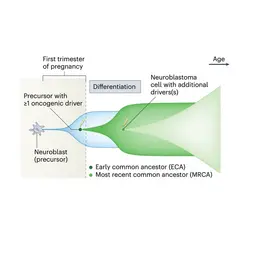
Bild: Computergestützte Rekonstruktion der Evolution von Neuroblastomen durch Genomsequenzierung (Schema adaptiert aus G. Caravagna, Mathematical modeling of neuroblastoma associates evolutionary patterns with outcomes, Nat. Genet. 2023)., © dkfz.de
Unsere Forschung

Aktuelle Forschungsschwerpunkte
Da krebsassoziierte Mutationen kontinuierlich in erneuernden Geweben wie dem Blut auftreten, ist es wahrscheinlich, dass diese Gewebe über Schutzmechanismen verfügen, die die Selektion von Krebs treibenden Mutationen und die maligne Transformation begrenzen. Um die Natur dieses Schutzes zu verstehen, untersuchen wir die Dynamik der nicht-malignen, homöostatischen und stressinduzierten Hämatopoese bei Mäusen und Menschen – sowohl auf der Ebene einzelner Zellklone (Busch et al., 2015; Pei et al., 2017) als auch hinsichtlich der zugrunde liegenden molekularen Regulation. Darüber hinaus erforschen wir mit Mausmodellen sporadischer Leukämien sowie menschlichen Knochenmark- und Blutproben, wie Störungen der normalen Stamm- und Vorläuferzelldynamik zur Entwicklung akuter Leukämien führen. Methoden zur Untersuchung der somatischen Evolution beim Menschen, insbesondere mithilfe der Populationsgenetik somatischer Mutationen, können auf solide Gewebe übertragen werden. Dadurch können wir die Evolution von Krebs im sich entwickelnden und erwachsenen Nervensystem untersuchen (Körber et al., 2023). Unsere Forschung zu klonalen Dynamiken in der somatischen Evolution begann mit Studien zur T-Zell-Antwort auf Einzelklonebene. Hier entwickelten wir ein mathematisches Inferenzmodell, das auf Basis experimenteller Daten vorhersagte, dass sich Gedächtnis-T-Zellen bereits früh während einer akuten Immunantwort aus einer stammzellähnlichen T-Zell-Subpopulation entwickeln (Buchholz et al., 2013). Diese Sichtweise hat mittlerweile breite Akzeptanz gefunden, und wir untersuchen weiterhin die klonale Dynamik adaptiver Immunantworten und deren Regulation. Alle unsere Projekte entstehen in enger Zusammenarbeit mit Experimentatoren und Theoretikern.
Aktuelle Entwicklungen
Die computergestützte Rekonstruktion der somatischen Evolution aus Genomsequenzierungsdaten ermöglicht uns die zeitliche Bestimmung zentraler genetischer Ereignisse in der Krebsentstehung. In Zusammenarbeit mit Forschern des Hopp-Kindertumorzentrums erforschen wir derzeit die Anwendung dieser Methoden zur Früherkennung von pädiatrischen Krebserkrankungen. Darüber hinaus entwickeln wir robuste Methoden zur Detektion klonaler Selektion in normalen Geweben.
Methoden und Technologien
Wir nutzen ein breites Spektrum an Methoden zur mathematischen Modellierung klonaler Dynamiken und der somatischen Evolution sowie zur Validierung dieser Modelle anhand experimenteller Daten. Hierbei setzen wir unter anderem auf Bayessche Inferenzmethoden und künstliche Intelligenz. Für in-situ Barcoding-Studien an Mäusen haben wir eine Reihe von computergestützten Werkzeugen entwickelt, die speziell auf die Polylox- und PolyloxExpress-Systeme zugeschnitten sind (Pei et al., 2017).
Ziele und gesellschaftliche Relevanz
Wir erwarten, dass das Verständnis der Mechanismen, die es prämalignen Klonen ermöglichen, in normalen Geweben zu wachsen und schließlich zu voll entwickelten malignen Tumoren zu werden, neue Methoden zur frühzeitigen Krebsdiagnose hervorbringen wird. Dies würde eine frühere und wirksamere Behandlung ermöglichen.
Projekte
Stammzelldynamik in Entwicklung und Homöostase
Die Differenzierungsraten von Stamm- und Vorläuferzellen sowie ihre Regulation in der Entwicklung und Homöostase können nicht direkt gemessen werden. Wir entwickeln computergestützte Methoden, um diese aus Barcoding-Daten bei Mäusen oder aus somatischen Variantenstatistiken beim Menschen abzuleiten. Unser Fokus liegt auf dem hämatopoetischen System, sowohl auf der definitiven Hämatopoese als auch auf der Entwicklung von Gewebsmakrophagen.
Somatische Evolution
Wir entwickeln Methoden zur Rekonstruktion der somatischen Evolution einzelner Gewebe. Diese wenden wir an, um die evolutionäre Dynamik von Krebs patientenspezifisch zu rekonstruieren. In jüngster Zeit konzentrieren wir uns zudem auf die Untersuchung von Mutations- und Selektionsdynamiken in normalen oder prämalignen Geweben.
Molekulare Regulation hämatopoetischer Stammzellentscheidungen
In enger Verbindung mit Projekt 1 (Stammzelldynamik) erforschen wir genregulatorische Netzwerke, die die Modi und Raten der Differenzierung und Proliferation hämatopoetischer Stamm- und Vorläuferzellen steuern.
Klonale Dynamik von Immunantworten
T-Zell-Immunantworten werden durch kleine Subpopulationen stammzellähnlicher T-Zellen aufrechterhalten. In kollaborativen Projekten untersuchen wir die molekulare Regulation der Schicksalsentscheidungen zwischen Stamm- und Effektorzellen. Darüber hinaus erforschen wir die klonale Dynamik von NK-Zell-Antworten.
Team
-

Prof. Dr. Thomas Höfer
-

Dr. Nils Benjamin Becker
-

Dr. Sarah Benedetto
-
Diana Best
-

Dr. Patrick Binder
-

Nina Claudino
-

Franziska Esau
-
Alessandro Greco
-
Dr. Matthias Günther
-
Philipp Höflich
-
Tatsuro Ikeda
-
Dr. Nikolaus Kepper
-
Emma Kray
-

Maurice Langhinrichs
-
Odette Luber
-

Jonas Metz
-

Tamar Nizharadze
-

Srishti Patil
-
Dr. Giada Sandrini
-
Aisha Schuhegger
-

Dr. Lea Weber
-
Meike Witschel
Ausgewählte Publikationen
Körber V et al.
Körber V et al.
Pei W, Feyerabend TB, et al.
Busch K et al.
Buchholz VR, Flossdorf M, et al.
Kontaktieren Sie uns

