Viele Gene kommen in verschiedenen Menschen in verschiedenen Varianten vor. Um solche Varianten zu entdecken, die das Risiko für eine bestimmte Krankheit erhöhen, vergleichen Wissenschaftler die Gene von Patienten mit denen von gesunden Kontrollpersonen. Doch häufig liefern diese Untersuchungen keine eindeutigen Ergebnisse: Denn der Effekt von bestimmten Erbgutvarianten hängt oft davon ab, ob auch andere Gene betroffen sind. Erst das Zusammenwirken von verschiedenen Genen hat Konsequenzen. Die nun von Boutros und Huber vorgestellte Methode kann diese Kombinationseffekte aufdecken.
Mit der so genannten RNA-Interferenz schalteten sie Gene einzeln und in allen paarweisen Kombinationen aus. Indem die Forscher systematisch alle Wechselwirkungen zwischen wichtigen Signalmolekülen katalogisierten, erhielten sie für jedes Gen eine detaillierte Liste von Interaktionspartnern, vergleichbar mit einer “Freundes-Liste" im sozialen Netzwerk “Facebook".
„Wenn zwei Nutzer von Facebook die gleichen Freunde haben, kann man mit hoher Wahrscheinlichkeit davon ausgehen, dass die beiden sich kennen – auch dann, wenn sie selbst nicht “Facebook-Freunde" sind“, erklärt Michael Boutros. „Übertragen auf die Situation im Erbgut kann man durch den Vergleich ihrer Wechselwirkungen vorhersagen, welche Gene eine gemeinsame Funktion ausüben.“
Boutros, Huber und ihre Kollegen können nun also “Freunde vorschlagen", Gene, die sich in ihrer Wirkung beeinflussen. Zum Beispiel entdeckten die Forscher bei ihren Experimenten eine bislang unbekannte Komponente des RAS-Signalweges, der bei der Entstehung von Krebs eine wichtige Rolle spielt. Diese neue Methode könnte deshalb dazu beitragen, neue Komponenten krebsrelevanter Signalketten zu finden und damit mögliche Angriffspunkte für neue Krebstherapien.
Diese Studien wurden vom Exzellenzcluster CellNetworks, in dem Boutros und Huber Mitglieder sind, maßgeblich gefördert.
Originalveröffentlichung: Horn, T., Sandmann, T., Fischer, B., Axelsson, E., Huber, W. & Boutros, M. Mapping of Signalling Networks through Synthetic Genetic Interaction Analysis by RNAi. Nature Methods, Advance Online Publication 6 March 2011. DOI: 10.1038/nmeth.1581.
Ein Bild zur Pressemitteilung steht im Internet zur Verfügung unter:
www.dkfz.de/de/presse/pressemitteilungen/2011/images/bild_pm_13.jpg
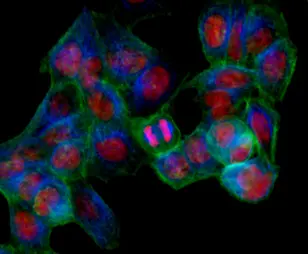
Bildunterschrift: Gene befinden sich auf der Erbsubstanz im Zellkern, auf diesem Foto sind die Zellkerne rot angefärbt.
Über das DKFZ
Das Deutsche Krebsforschungszentrum (DKFZ) ist mit mehr als 3.000 Mitarbeiterinnen und Mitarbeitern die größte biomedizinische Forschungseinrichtung in Deutschland. Wissenschaftlerinnen und Wissenschaftler erforschen im DKFZ, wie Krebs entsteht, erfassen Krebsrisikofaktoren und suchen nach neuen Strategien, die verhindern, dass Menschen an Krebs erkranken. Sie entwickeln neue Methoden, mit denen Tumoren präziser diagnostiziert und Krebspatienten erfolgreicher behandelt werden können. Beim Krebsinformationsdienst (KID) des DKFZ erhalten Betroffene, Interessierte und Fachkreise individuelle Antworten auf alle Fragen zum Thema Krebs.
Um vielversprechende Ansätze aus der Krebsforschung in die Klinik zu übertragen und so die Chancen von Patientinnen und Patienten zu verbessern, betreibt das DKFZ gemeinsam mit exzellenten Universitätskliniken und Forschungseinrichtungen in ganz Deutschland Translationszentren:
- Nationales Centrum für Tumorerkrankungen (NCT, 6 Standorte)
- Deutsches Konsortium für Translationale Krebsforschung (DKTK, 8 Standorte)
- Hopp-Kindertumorzentrum (KiTZ) Heidelberg
- Helmholtz-Institut für translationale Onkologie (HI-TRON) Mainz – ein Helmholtz-Institut des DKFZ
- DKFZ-Hector Krebsinstitut an der Universitätsmedizin Mannheim
- Nationales Krebspräventionszentrum (gemeinsam mit der Deutschen Krebshilfe)
Das DKFZ wird zu 90 Prozent vom Bundesministerium für Bildung und Forschung und zu 10 Prozent vom Land Baden-Württemberg finanziert und ist Mitglied in der Helmholtz-Gemeinschaft Deutscher Forschungszentren.