Cell Fate Engineering and Disease Modeling
- Cell and Tumor Biology
- Junior Research Group

Dr. Moritz Mall
One of the most exciting concepts in biology is the plasticity of cell fate that allows cellular identity to be reset. Strikingly, this plasticity is essential for normal development, but several human diseases are also associated with unwanted changes in cell identity.
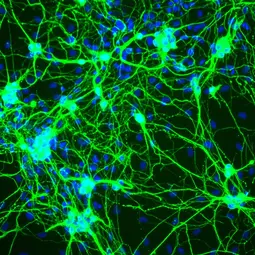
Our Research
Dedifferentiation and adoption of stem cell-like properties for example are hallmarks of cancer and aberrant gene expression is linked to neuropsychiatric diseases. Investigating the mechanisms that safeguard cell identity will therefore provide new opportunities to understand and treat these devastating diseases.
Cell fate engineering enables researchers to change the identity of cells from one type to another. It allows for example to take blood or skin cells from a patient suffering autism and to reprogram them into functional human neurons in order to study the disease in the culture dish. This revolutionizing technology offers a novel platform to analyze cell identity and disease in cells that are normally not available, such as patient brain cells (Mall and Wernig, 2017; Yang, 2017). Our work showed that active repression of unwanted genetic programs is important to safeguard the identity of neurons (Treutlein, Nature 2016; Mall, Nature 2017). Since the factors involved are also linked to mental disorders and brain malignancies we will investigate the exciting possibility how loss of neuronal identity can contribute to these devastating diseases.
Projects
Universitäre KI-Forschung zur Entwicklung einer Software als Medizinprodukt für die klinische Patientenversorgung am Beispiel eines Assistenzsystems für die Hautkrebsdiagnostik
Weltweit wurden im Jahr 2020 rund 325.000 Fälle des Melanoms diagnostiziert, ca. 60.000 Menschen sind letztlich daran gestorben. Während einige Melanome bereits im Frühstadium ein aggressives Verhalten zeigen, steigt die Wahrscheinlichkeit einer Metastasierung mit zunehmender Tumordicke an. Folglich ist eine schnelle und präzise Identifizierung des Melanoms von immenser Bedeutung.
Die Frühdiagnose ist jedoch selbst für erfahrene DermatologInnen nicht einfach, da sich Melanome und atypische Nävi häufig morphologisch überschneiden. Die diagnostische Herausforderung, Melanome frühzeitig zu erkennen und dabei gleichzeitig die Überdiagnose (Falsch-Positiv-Rate) zu minimieren, erfordert daher die Entwicklung fortschrittlicher Diagnosesysteme. In diesem Kontext zeigten insbesondere sogenannte Deep Neural Networks bei der Klassifizierung verdächtiger dermatoskopischer Bildaufnahmen vergleichbare oder sogar bessere Ergebnisse als erfahrene DermatologInnen.
Was bislang jedoch fehlt, ist die Translation dieser vielversprechenden Forschungsergebnisse in die klinische Routine, um einen realen Nutzen für PatientInnen, ÄrztInnen und das Gesundheitssystem zu schaffen. Vor diesem Hintergrund wird im Rahmen des sKIn-Projekts modellhaft ein auf künstlicher Intelligenz (KI) basierendes Assistenzsystem für die Melanomdiagnostik weiterentwickelt une unter Berücksichtigung der europäischen Medizinprodukteverordnung (MDR) aus der universitären Forschung zur Marktfähigkeit gebracht. Diese erklärbare KI wird weiterhin gemeinsam mit einem renommierten Dermatoskophersteller in digitale Dermatoskope integriert, um so flächendeckend eine Integration in die Hautkrebsscreening-Untersuchungen zu ermöglichen. Auf diese Weise wird eine Verbesserung der Melanomdiagnostik und somit realer Mehrwert für PatientInnen, ÄrztInnen sowie das Gesundheitssystem geschaffen.
Gleichzeitig dient das sKIn-Projekt als Blaupause für andere Forschungseinrichtungen und soll perspektivisch die Translation KI-basierter Software erleichtern. Hierfür werden konkrete Handlungsempfehlungen erarbeitet und Anregungen für die weitere Ausgestaltung regulatorischer und gesundheitspolitischer Rahmenbedingungen aufbereitet.
Heterogenität, Entwicklung und Resistenz von fusionsbedingten Sarkomen bei Jugendlichen und jungen Erwachsenen (BMBF)
Tumore entwickeln sich durch genetische und nicht-genetische Veränderungen, die zu intra-tumoraler Heterogenität (ITH) führen, einem Schlüsselfaktor für Rückfälle und Therapieresistenz. Unser Schwerpunkt liegt auf Knochen- und Weichteilsarkomen, die durch chimäre Fusionsgene verursacht werden. Diese dienen als Modellkrankheiten für die Untersuchung von ITH bei Krebserkrankungen mit definierten genetischen Triebkräften, um die Erkenntnisse in innovative Behandlungsstrategien gegen verschiedene und sich entwickelnde Tumore umzusetzen. Zu den Herausforderungen gehören begrenzte wirksame Therapien, stagnierende Behandlungsergebnisse und ein frühes Auftreten, was die Dringlichkeit unterstreicht, die Behandlungsergebnisse von Sarkom-Patienten zu verbessern. Fusionsgetriebene Sarkome (FDS) bieten aufgrund ihrer entitätsbestimmenden Fusionsgene eine einzigartige Gelegenheit, die Untersuchung von Subklonen und Plastizität zu erleichtern. FDS weisen auch eine geringe genetische Komplexität auf, was die Identifizierung von ITH-fördernden Faktoren und das Ansprechen auf eine Therapie erleichtert. Wir nutzen das Fachwissen von HEROES-AYA, einem internationalen Netzwerk für Präzisionsonkologie, und wollen FDS bei der Diagnose und während der Therapieresistenz durch modernste Analysen auf Einzelzell- und räumlicher Ebene untersuchen. In-vitro- und In-vivo-Validierungen von Schwachstellen werden in die Entwicklung von klinischen Studien über alle Altersgruppen hinweg einfließen und eine neue Ära der Präzisionsonkologie in Deutschland einläuten. Die Gruppe trägt wesentlich zu den datenwissenschaftlichen Aspekten dieses Projekts bei.
Eine Deep-Learning-Strategie für die Hochdurchsatz-Proteomik von Blutplasma und einzelnen Zellen (Heidelberg Explorer Call)
Dieses Projekt ist eine Kooperation mit dem Proteomik-Labor von Prof. Krijgsveld. Proteine sind die wichtigsten Triebkräfte für Zellfunktionen und Krankheiten. Daher ist die Proteomik ein sehr geeignetes Verfahren zur Charakterisierung von Determinanten der Zellidentität und zur Ermittlung von Biomarkern. Die derzeitige Proteomik-Technologie ist so umfangreich, dass sie Profile von Tausenden von Proteinen erstellen kann, und so empfindlich, dass sie auf einzelne Zellen zugreifen kann. Allerdings fehlt ihr der Durchsatz für eine aussagekräftige Analyse großer Probenkohorten sowohl in der Grundlagenforschung als auch in der Klinik. Daher entwickeln wir neuartige, auf künstlicher Intelligenz (KI) basierende Ansätze für die Analyse massenspektrometrischer (MS) Daten, um Plasmaproteomdaten dem klinischen Status zuzuordnen und Einzelzelldaten mit der Zellidentität zu korrelieren. Darüber hinaus werden wir Proteom-Experimente durchführen, um zu zeigen, dass dieser auf Deep Learning basierende Ansatz den Durchsatz der Plasma- und Einzelzell-Proteomanalyse erhöht. Insgesamt wird dieses Projekt einen bahnbrechenden Ansatz in der Proteomik einführen, der die Stratifizierung von Patienten und die Entdeckung von Biomarkern ermöglicht und das junge Feld der Einzelzell-Proteomik voranbringt.
Entwicklung von digitalen Biomarkern für BAP1-mutierte Krebsarten (Deutsche Krebshilfe)
Im Mittelpunkt des Projekts steht BAP1, ein Tumorsuppressor-Gen, das bei aggressiven Krebsarten häufig mutiert ist. Insbesondere BAP1-Mutationen führen zu einer erhöhten Aggressivität und Metastasierung des Tumors. Das Ziel dieses Projekts ist es, die Anfälligkeit von BAP1-Mutationen zu verstehen und sie für potenzielle Therapien bei BAP1-mutierten Krebsarten zu nutzen. Um dies zu erreichen, wurde ein kollaboratives Konsortium gebildet, dessen Teilprojekte sich auf die Bereitstellung der erforderlichen Materialien, die Prüfung spezifischer Medikamente in vitro, in vivo und in von Patienten stammenden Organoiden sowie auf die Durchführung von Biomarker-Analysen an großen menschlichen Gewebekohorten konzentrieren. Das übergeordnete Ziel besteht darin, den vorgeschlagenen Mechanismus zu validieren und potenzielle Behandlungsmöglichkeiten zu erforschen, wobei der Schwerpunkt auf der Einleitung klinischer Versuche auf der Grundlage der Ergebnisse liegt. Das Projekt integriert verschiedene Ansätze, darunter In-vitro-, In-vivo-, Ex-vivo- und klinische Probenanalysen, um BAP1-mutierte Krebsarten umfassend zu untersuchen und Strategien für eine personalisierte Medizin zu entwickeln. Innerhalb dieses Konsortiums ist unsere Gruppe für die Entwicklung digitaler Biomarker zur besseren Stratifizierung von Patienten zuständig.
Team
A team of 20 people from the fields of medicine, molecular biology and computer science / data science focuses on the identification of relevant patterns in patient data as well as increased explainability and security of artificial intelligence decisions.
- Show profile

Dr. Moritz Mall
-
Annasara Artioli
-
Diana Avetyan
-
Hanna Dorothea Belschner
-
Matteo Gasparotto
-
Francesco Giannone
-

Sarah Grieder-Mall
-
Catello Guida
-
Sebastian Heine
-
Anne Hoffrichter
-
Dr. Sandra Horschitz
-

Sandra Keßler
-

Tim Kindinger
-
Louis Masclef
-
Raquel Perez Fernandez
-
Laura Rueda Gensini
-
Nathan Trausch
-
Jule Truberg
Future Outlook
We mainly employ pluripotent stem cells and cell fate engineering to reconstruct and investigate human development and disease. Our mission is to understand the mechanisms that determine and maintain cell fate with the goal to treat diseases associated with loss of cell identity. Our immediate research focus is to understand the role of cell identity loss in brain malignancies and mental disorders that affect millions of patients worldwide and are a major medical and economic challenge to our modern society.

Selected Publications
MEOX2 homeobox gene promotes growth of malignant gliomas
A. Schönrock*, E. Heinzelmann*, B. Steffl, E. Demirdizen, A. Narayanan, D. Krunic, M. Bähr, J.W. Park, C. Schmidt, K. Özduman, M.N. Pamir, W. Wick, F. Bestvater, D. Weichenhan, C. Plass, J. Taranda, M. Mall, S. Turcan.
Trumpp, A. and S. Haas
Pro-neuronal activity of Myod1 due to promiscuous binding to neuronal genes
Q.Y. Lee*, M. Mall*, S. Chanda, B. Zhou, K.S. Sharma, K. Schaukowitch, J.M. Adrian-Segarra, S.D. Grieder, M.S. Kareta, O. Wapinski, C.E. Ang, R. Li, T.C. Südhof, H.Y. Chang, M. Wernig
Get in touch with us


