Strukturbiologie von Infektion und Immunität
- Immunologie, Infektion und Krebs

Dr. Erec Stebbins
Head of Division/Abteilungsleiter
Unsere Forschung beschäftigt sich mit der mechanistischen Beschreibung des Erregers der Afrikanischen Schlafkrankheit (Trypanosoma brucei). Wir untersuchen dabei insbesondere das auf seiner Oberfläche präsente und als Antigen wirkende variable Oberflächenglykoprotein (variant surface glycoprotein). Durch seine einzigartigen immunologischen Eigenschaften dient uns T. brucei als Modellsystem, um zu verstehen, wie sich Infektions- und Krebserkrankungen der Erkennung durch das Immunsystem entziehen.
Bild: Links: Bilder von Trypanosoma brucei, die sich im Blut vermehren; unten ist seine Protein-Oberflächenstruktur zu erkennen, während es von einem VSG zum nächsten wechselt. Rechts oben: Proteinkristall und Beugungsmuster. Rechts unten: Struktur von VSGsur, gebunden an das Anti-Trypanosomen-Medikament Suramin.,
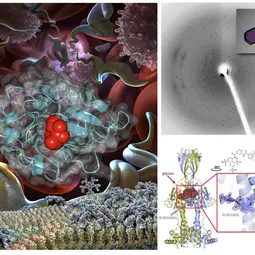
Bild: Links: Bilder von Trypanosoma brucei, die sich im Blut vermehren; unten ist seine Protein-Oberflächenstruktur zu erkennen, während es von einem VSG zum nächsten wechselt. Rechts oben: Proteinkristall und Beugungsmuster. Rechts unten: Struktur von VSGsur, gebunden an das Anti-Trypanosomen-Medikament Suramin.,
Unsere Forschung
Unsere Forschung
Verschiedene Formen der Immunevasion sind typisch für die langfristige Pathogenese, sowohl bei Infektionskrankheiten als auch bei Krebserkrankungen. Das Immunsystem erkennt fremde und gefährliche „eigene“ Antigene und beseitigt Zellen die sie beherbergen, wie Mikroben und Tumore. Krankheitserreger und behandlungsresistente Krebserkrankungen wirken der Reaktion des Immunsystems oft entgegen, indem sie ihre Oberflächenantigene verändern. Wir verwenden ein kontrolliertes, biologisch sicheres, genetisch manipulierbares Modellsystem (afrikanische Trypanosomen der Spezies T. brucei), um langfristige, immuninvasive Prozesse im Rahmen des kooperativen, starken Immunologie_Forschungsprogramms des Deutschen Krebsforschungszentrums zu untersuchen. Unsere ersten Forschungsergebnisse haben bereits einen Paradigmenwechsel bewirkt.
T. brucei verursacht die afrikanische Schlafkrankheit beim Menschen und eine verwandte Erkrankung in Tieren. Das sogenannte „Variant Surface Glycoprotein“ (VSG) des Erregers spielt dabei eine zentrale Rolle, da der Erreger damit dem Angriff des menschlichen Immunsystems entgehen kann. Auf der Oberfläche des Erregers bildet dieses VSG eine einzigartige und extrem dichte Hülle aus etwa 10 Millionen Molekülen. Das Immunsystem erkennt diese Oberflächenhülle als gefährliches Antigen und reagiert entsprechend mit einer starken Antikörperreaktion. Der Parasit entzieht sich diesem Angriff allerdings, indem er auf ein großes genetisches Repertoire abweichender VSG zugreifen, und seine Oberflächenstruktur quasi auf eine neue (unterschiedliche Antigene präsentierende) VSG-Variante „umschalten“ kann. Dies führt zu einer langanhaltenden Infektion, in deren Verlauf die Parasitenbelastung immer wieder zwischen Höchst- und Tiefstwerten schwankt. Diese Schwankungen sind das Ergebnis eines Prozesses, der als Antigenvariation bekannt ist, und der sich durch wiederholte Zyklen von Antikörperbildung, Abtötung der Parasiten und erneuter Parasitenbelastung nach dem Wechsel der VSG-Oberfläche auszeichnet.
Unsere Forschung zielt darauf ab, die sehr unterschiedlichen VSG-Proteine und ihre Wechselwirkung mit den Antikörpern mechanistisch zu charakterisieren. Unser „Bottom-Up“-Ansatz mit Hilfe der Röntgenkristallographie hat unsere Vorstellung davon, wie T. brucei es schafft sich dem Immunsystem zu entziehen dramatisch verändert. Bedeutende Erfolge waren dabei (1) die Entdeckung unerwarteter und stark ausgeprägter struktureller Abweichungen zwischen den unterschiedlichen VSGs, (2) die Identifizierung unvorhergesehener und immunmodulatorisch wirkender, post-translationaler Modifikationen der VSGs, die das „Epitop-Spektrum“ erheblich erweitern, (3) die Entdeckung von Co-Kristallstrukturen zwischen den Antikörpern und der VSG, die erstmals eine Erfassung der Epitop-Verfügbarkeit auf der Oberflächenhülle ermöglichte, (4) der Nachweis von VSG-Funktionen, die über reine Antigen-Variation hinausgehen (z.B. die Bindung an biologisch relevante Substrate), (5) der Nachweis, dass die einzelnen VSGs zur Ausschüttung ungewöhnlicher Antikörperrepertoires führen, die sehr genau auf die immundominanten Epitope abzielen, und (6) die translationale Entwicklung einer neuartigen und leistungsfähigen Impfstoff-Plattform, die auf diesem Oberflächenmantel basiert und entscheidend durch die Erkenntnisse aus der Erforschung seiner Proteinstruktur geprägt ist.
Vor diesem Hintergrund arbeiten unsere Techniker:innen, Student:innen und Wissenschaftler:innen an verschiedenen Projekten zur Charakterisierung der unterschiedlichen VSG-Proteine (und erfassen dabei nach und nach deren volle immunologische Vielfalt) und der Co-Kristallstrukturen zwischen Antikörpern und Antigenen. Zu den dabei eingesetzten Labortechniken und Methoden gehören die Anlage von Gewebekulturen, die Kultivierung eukaryotischer Mikroorganismen, die Aufreinigung und Kristallisation von Proteinen im großen Maßstab (wir arbeiten sowohl mit den endogenen Proteinen der afrikanischen Trypanosomen als auch mit rekombinanten Proteinen, die mittels bakterieller Expressionsverfahren gewonnen werden), die Strukturbestimmung mit Hilfe der Röntgenkristallographie sowie die Kryoelektronenmikroskopie.
Fotos des Labors und der Aktivitäten
-

Bild von mehreren VSG-Strukturen -

DKFZ-Gebäude auf dem Campus der Universität Heidelberg -

Lab und Freunde auf dem Weihnachtsmarkt in Heidelberg. -

Röntgendiffraktometer am DKFZ -

Röntgendiffraktometer am DKFZ -

Kryokühlung mit flüssigem N2 für Röntgendiffraktometer am DKFZ -

Röntgenbeugungsbild -

Laborraum -

Bilder aus der Veröffentlichung -

Bilder aus der Veröffentlichung -

Personen und Ergebnisse der Veröffentlichung -

DKFZ-Hauptgebäude auf dem Campus der Universität Heidelberg -

Labor-BBQ-Ausflug in der Grillhütte in Heidelberg -

Bilder aus der Veröffentlichung -

Besuch von Direktor Baumann -

Bilder aus der Veröffentlichung -

VAST-Immuntherapeutika-Plattform -

VAST-Immuntherapeutika-Plattform -

Laborausflug im Ketterwald (Baumklettern und Hindernislauf) -

4,5 Mio. EURO Bundeszuschuss an DKFZ und Ausgründung Panosome
Team
-

Dr. Erec Stebbins
Head of Department/Abteilungsleiter
-

Dr. Tanja Isabell Kuhm
Postdoc
-

Caterina Canetta
Doktorand
-

Johan Philip Zeelen
Technischer Ingenieur
-

Monique van Straaten
Technischer Ingenieur
- Profil anzeigen

Christoph Tim Pufall
Administrative Assistant
Ausgewählte Publikationen
Dakovic S, Zeelen JP, Gkeka A, Chandra M, van Straaten M, Foti K, Zhong J, Vlachou EP, Aresta-Branco F, Verdi JP, Papavasiliou FN, Stebbins CE
Gkeka A, Aresta-Branco F, Triller G, Vlachou EP, van Straaten M, Lilic M, Olinares PDB, Perez K, Chait BT, Blatnik R, Ruppert T, Verdi JP, Stebbins CE, Papavasiliou FN
Chandra M, Dakovic S, Foti K, Zeelen JP, van Straaten M, Aresta-Branco F, Tihon E, Lübbehusen N, Ruppert T, Glover L, Papavasiliou FN, Stebbins CE
Triller G, Vlachou EP, Hashemi H, van Straaten M, Zeelen JP, Kelemen Y, Baehr C, Marker CL, Ruf S, Svirina A, Chandra M, Urban K, Gkeka A, Kruse S, Baumann A, Miller AK, Bartel M, Pravetoni M, Stebbins CE, Papavasiliou FN, Verdi JP
Kontaktieren Sie uns
